ChIP-seq je úžasná technika, která nám umožňuje zkoumat fyzikální vazebné interakce mezi proteinem a DNA pomocí sekvenování nové generace. V tomto článku podám stručný přehled ChIP a představím techniku sekvenování chromatinové imunoprecipitace (ChIP-seq), která kombinuje ChIP se sekvenováním nové generace.
Co je chromatinová imunoprecipitace?
Chromatinová imunoprecipitace (ChIP) nám umožňuje určit vazebná místa proteinů na DNA. Chromatin je komplex DNA zabalený s histonovými proteiny do nukleozomů. ChIP využívá reverzibilních příčných vazeb vytvořených mezi DNA a přidruženými proteiny při formaldehydové fixaci buněk nebo tkáně. Fixovaný chromatin je fyzicky střižen a fragmenty DNA spojené s určitým proteinem jsou selektivně imunoprecipitovány a analyzovány. Analýza může probíhat lokus po lokusu pomocí PCR, ale častěji se ChIP zkoumá pomocí mikročipů (ChIP-chip) nebo sekvenování nové generace (ChIP-seq).
Jak funguje ChIP-chip?
ChIP-on-chip neboli ChIP-chip kombinuje imunoprecipitaci chromatinu s analýzou mikročipů (čipů). V roce 2001 publikoval Jason Lieb z laboratoře Pata Browna ve Stanfordu první článek o ChIP-chip (Lieb 2001). Při této metodě se fragmenty DNA, které se vysrážejí s určitým proteinem, nanesou na mikročip pro analýzu. Tím se získá globální obraz o tom, kde se protein váže, na rozdíl od pouhého zkoumání jednotlivých vazebných míst pomocí PCR. Přestože se jednalo o revoluční přístup, je tato technika omezena použitými maticovými technologiemi. Zaprvé, potřebujete mikroarray pro genom, který chcete studovat, a to na začátku až v polovině roku 2000 často nebylo. Za druhé, tyto mikročipy jsou omezeny kvalitou referenčního genomu a schopností navrhnout sondy, které budou na poli fungovat. Za třetí, matrice jsou omezené v tom, jak velkou část genomu mohou reprezentovat, a obvykle se rozprostírají napříč genomem s rozlišením 100 nebo 1000 bp. Existují také problémy týkající se zkreslení amplifikace fragmentů DNA ChIP, normalizace dat array a srovnatelnosti platforem array.
Jak funguje ChIP-seq?
Chromatinové imunoprecipitační sekvenování neboli ChIP-seq kombinuje ChIP se sekvenováním nové generace (Barski 2007, Johnson 2007). Protokoly ChIP-seq byly převzaty z metod ChIP-chip: proteiny jsou zesíťovány s navázanou DNA ošetřením formaldehydem, buňky jsou homogenizovány a chromatin je střižen a imunoprecipitován magnetickými kuličkami navázanými na protilátky. Imunoprecipitovaná DNA se poté použije jako vstup pro protokol přípravy knihovny sekvenování nové generace, kde se sekvenuje a analyzuje na vazebná místa DNA. Shrnutí pracovního postupu ChIP-seq a příklad výsledků ChIP-seq viz obrázek níže (reprodukováno s laskavým svolením Dominica Schmidta (Schmidt 2009)).
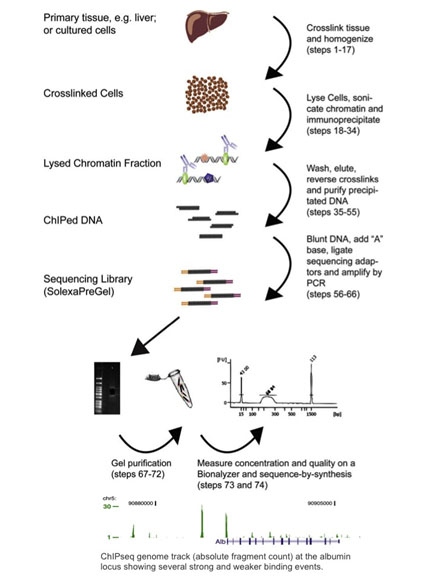
Ačkoli většina z přibližně 400 dosud publikovaných prací byla analyzována na platformě Illumina, ChIP-seq lze provádět na jakémkoli sekvenátoru nové generace (Wold 2008). ChIP-seq se široce rozšířil od doby, kdy byl poprvé popsán v roce 2007. Ve skutečnosti téměř zcela vytlačil ChIP-Chip, protože umožňuje analýzu celého genomu a nemá výše uvedená omezení.
Co lze vlastně s ChIP-seq dělat
ChIP-seq je mocný a všestranný nástroj a v literatuře existuje mnoho skvělých příkladů použití ChIP-seq. Vybral jsem několik svých oblíbených (z prací prováděných v základním zařízení, které řídím), abych ilustroval, co je možné, a zahrnul příklady, kdy ChIP-seq inspiroval vývoj nových metod:
- Antoni Hurtado a kol. provedli knock-down “pionýrského faktoru” FoxA1, což vedlo ke snížení vazby estrogenového receptoru (ER) na více než 50 % známých vazebných míst ER. Ukázali, že FoxA1 je důležitým regulátorem transkripce zprostředkované ER, což naznačuje, že může být novým a důležitým terapeutickým cílem u rakoviny prsu (Hurtado 2011).
- Dominic Shmidt, et al. použili ChIP-seq ke zkoumání vývoje vazby transkripčních faktorů. Zaměřili se na vazbu CEBPA a HNF4 v jaterní tkáni pěti druhů obratlovců: člověka, myši, psa, vačice a kuřete. Metoda ChIP-chip by byla téměř nemožná vzhledem k rozdílným druhům a složitosti návrhu sond (Schmidt 2010).
Další modifikace metody ChIP-seq vedly k několika novým metodám, včetně ChIP-seq pro analýzu interakcí mezi RNA a proteiny a také DNase-seq a FAIRE-seq, které se používají k identifikaci regulačních oblastí v DNA.
Shrnem lze říci, že ChIP-seq je vyspělá technika, která je vhodnější než ChIP-chip a kterou může používat téměř každá skupina se zájmem o analýzu interakcí DNA:protein. Jak byste mohli využít ChIP-seq ve svém výzkumu?
Barski et al, High-resolution profiling of histone methylations in the human genome (Profilování histonových metylací v lidském genomu s vysokým rozlišením). Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promotor-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Aplikace technologií sekvenování nové generace ve funkční genomice. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Pomohlo vám to? Pak se prosím podělte se svou sítí.
.