ChIP-seq ist eine wunderbare Technik, die es uns ermöglicht, die physikalischen Bindungsinteraktionen zwischen Protein und DNA mit Hilfe der Sequenzierung der nächsten Generation zu untersuchen. In diesem Artikel gebe ich einen kurzen Überblick über ChIP und stelle die Chromatin-Immunpräzipitations-Sequenzierungstechnik (ChIP-seq) vor, die ChIP mit der Sequenzierung der nächsten Generation kombiniert.
Was ist Chromatin-Immunpräzipitation?
Mit der Chromatin-Immunpräzipitation (ChIP) können wir Proteinbindungsstellen auf der DNA bestimmen. Chromatin ist der Komplex aus DNA, der mit Histonproteinen in Nukleosomen verpackt ist. ChIP nutzt die reversiblen Querverbindungen, die durch die Formaldehyd-Fixierung von Zellen oder Gewebe zwischen DNA und assoziierten Proteinen entstehen. Das fixierte Chromatin wird physikalisch abgeschert, und die mit einem bestimmten Protein assoziierten DNA-Fragmente werden selektiv immunpräzipitiert und analysiert. Die Analyse kann mittels PCR für jeden einzelnen Locus erfolgen, häufiger wird ChIP jedoch mit Microarrays (ChIP-Chip) oder Next-Generation-Sequencing (ChIP-seq) untersucht.
Wie funktioniert ChIP-Chip?
ChIP-on-chip oder ChIP-Chip kombiniert die Chromatin-Immunpräzipitation mit der Microarray-Analyse (Chip). Im Jahr 2001 veröffentlichte Jason Lieb im Labor von Pat Brown in Stanford die erste ChIP-Chip-Arbeit (Lieb 2001). Bei dieser Methode werden die DNA-Fragmente, die sich mit einem bestimmten Protein ausfällen, zur Analyse auf einen Microarray-Chip aufgebracht. Auf diese Weise entsteht ein globales Bild davon, wo das Protein bindet, im Gegensatz zur einfachen Abfrage einzelner Bindungsstellen mittels PCR. Dies war zwar ein revolutionärer Ansatz, aber die Technik ist durch die verwendeten Array-Technologien begrenzt. Erstens benötigt man einen Microarray für das zu untersuchende Genom, und das war Anfang bis Mitte der 2000er Jahre oft nicht der Fall. Zweitens sind diese Microarrays durch die Qualität des Referenzgenoms und die Fähigkeit, Sonden zu entwickeln, die auf einem Array funktionieren, begrenzt. Drittens können Arrays nur einen begrenzten Teil des Genoms abbilden, da sie in der Regel das gesamte Genom mit einer Auflösung von 100 oder 1000 bp abbilden. Außerdem gibt es Probleme mit der Verzerrung bei der Amplifikation der ChIP-DNA-Fragmente, der Normalisierung der Array-Daten und der Vergleichbarkeit der Array-Plattformen.
Wie funktioniert ChIP-seq?
Chromatin-Immunpräzipitations-Sequenzierung oder ChIP-seq kombiniert ChIP mit Sequenzierung der nächsten Generation (Barski 2007, Johnson 2007). ChIP-seq-Protokolle wurden von ChIP-Chip-Methoden übernommen: Proteine werden durch Formaldehydbehandlung an ihre gebundene DNA vernetzt, die Zellen werden homogenisiert, das Chromatin wird geschert und mit Antikörper-gebundenen magnetischen Kügelchen immunpräzipitiert. Die immunpräzipitierte DNA wird dann als Input für ein Protokoll zur Vorbereitung von Sequenzierbibliotheken der nächsten Generation verwendet, wo sie sequenziert und auf DNA-Bindungsstellen analysiert wird. Die folgende Abbildung zeigt eine Zusammenfassung des ChIP-seq-Arbeitsablaufs und ein Beispiel für ChIP-seq-Ergebnisse (mit freundlicher Genehmigung von Dominic Schmidt (Schmidt 2009)).
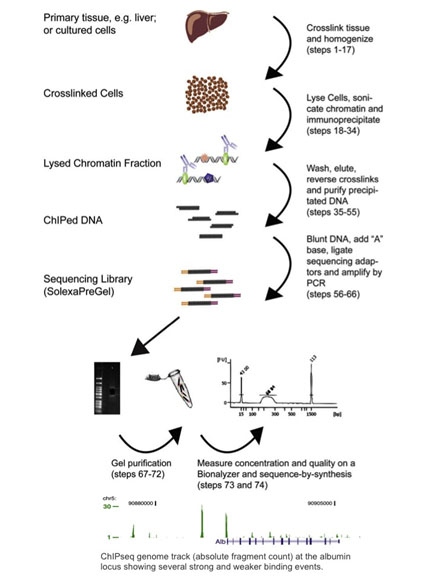
Obwohl die meisten der rund 400 bisher veröffentlichten Arbeiten auf der Illumina-Plattform analysiert wurden, kann ChIP-seq auf jedem Sequenzierer der nächsten Generation durchgeführt werden (Wold 2008). ChIP-seq hat sich seit seiner ersten Veröffentlichung im Jahr 2007 weitgehend durchgesetzt. Tatsächlich hat es den ChIP-Chip fast vollständig verdrängt, da es genomweite Analysen ermöglicht und die oben genannten Einschränkungen nicht aufweist.
Was kann man eigentlich mit ChIP-seq machen
ChIP-seq ist ein leistungsfähiges und vielseitiges Werkzeug, und es gibt in der Literatur viele großartige Beispiele für die Verwendung von ChIP-seq. Ich habe einige meiner Favoriten ausgewählt (aus Arbeiten, die in der von mir geleiteten Core Facility durchgeführt wurden), um zu veranschaulichen, was möglich ist, und Beispiele angefügt, bei denen ChIP-seq die Entwicklung neuer Methoden inspiriert hat:
- Antoni Hurtado et al. haben den “Pionierfaktor” FoxA1 ausgeschaltet, was zu einer verringerten Bindung durch den Östrogenrezeptor (ER) an über 50 % der bekannten ER-Bindungsstellen führte. Sie zeigten, dass FoxA1 ein wichtiger Regulator der ER-vermittelten Transkription ist, was darauf hindeutet, dass er ein neues und wichtiges therapeutisches Ziel bei Brustkrebs sein könnte (Hurtado 2011).
- Dominic Shmidt, et al. verwendeten ChIP-seq, um die Entwicklung der Bindung von Transkriptionsfaktoren zu untersuchen. Sie konzentrierten sich auf die Bindung von CEBPA und HNF4 im Lebergewebe von fünf Wirbeltierarten: Mensch, Maus, Hund, Opossum und Huhn. Ein ChIP-Chip wäre angesichts der unterschiedlichen Spezies und der Komplexität des Sondenentwurfs fast unmöglich gewesen (Schmidt 2010).
Weitere Modifikationen der ChIP-seq-Methode haben zu mehreren neuen Methoden geführt, darunter ChIP-seq für die Analyse von RNA-Protein-Interaktionen sowie DNase-seq und FAIRE-seq, die beide zur Identifizierung regulatorischer Regionen in der DNA verwendet werden.
Zusammenfassend lässt sich sagen, dass ChIP-seq eine ausgereifte Technik ist, die dem ChIP-Chip vorzuziehen ist und von fast jeder Gruppe eingesetzt werden kann, die an der Analyse von DNA-Protein-Interaktionen interessiert ist. Wie könnten Sie ChIP-seq in Ihrer Forschung einsetzen?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Applications of next-generation sequencing technologies in functional genomics. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Hat Ihnen das geholfen? Dann teilen Sie es bitte mit Ihrem Netzwerk.