ChIP-seq est une merveilleuse technique qui nous permet d’interroger les interactions de liaison physique entre la protéine et l’ADN en utilisant le séquençage de nouvelle génération. Dans cet article, je vais faire une brève revue de la ChIP et présenter la technique de séquençage par immunoprécipitation de la chromatine (ChIP-seq), qui combine la ChIP avec le séquençage de nouvelle génération.
Qu’est-ce que l’immunoprécipitation de la chromatine ?
L’immunoprécipitation de la chromatine (ChIP) nous permet de déterminer les sites de liaison des protéines sur l’ADN. La chromatine est le complexe d’ADN emballé avec des protéines histones en nucléosomes. La ChIP utilise les liaisons transversales réversibles établies entre l’ADN et les protéines associées par la fixation au formaldéhyde des cellules ou des tissus. La chromatine fixée est physiquement cisaillée et les fragments d’ADN associés à une protéine particulière sont sélectivement immunoprécipités et analysés. L’analyse peut se faire locus par locus à l’aide de la PCR, mais le plus souvent, la ChIP est interrogée à l’aide de microréseaux (ChIP-chip) ou de séquençage de nouvelle génération (ChIP-seq).
Comment fonctionne la ChIP-chip ?
La ChIP sur puce, ou ChIP-chip, combine l’immunoprécipitation de la chromatine avec l’analyse des microréseaux (puces). En 2001, Jason Lieb du laboratoire de Pat Brown à Stanford a publié le premier article sur la ChIP-chip (Lieb 2001). Dans cette méthode, les fragments d’ADN qui précipitent avec une protéine particulière sont appliqués sur une puce à ADN pour être analysés. On obtient ainsi une image globale de l’endroit où la protéine se lie, contrairement à la simple interrogation de sites de liaison uniques par PCR. Bien qu’il s’agisse d’une approche révolutionnaire, la technique est limitée par les technologies des puces utilisées. Tout d’abord, il faut disposer d’une biopuce pour le génome que l’on veut étudier, ce qui n’était souvent pas le cas au début ou au milieu des années 2000. Deuxièmement, ces puces sont limitées par la qualité du génome de référence et la capacité de concevoir des sondes qui fonctionnent sur une puce. Troisièmement, les réseaux sont limités quant à la quantité de génome qui peut être représentée, et ils sont généralement répartis sur le génome avec une résolution de 100 ou 1000 pb. Il y a également des problèmes de biais dans l’amplification des fragments d’ADN de ChIP, de normalisation des données de tableau et de comparabilité des plateformes de tableau.
Comment fonctionne ChIP-seq ?
Le séquençage par immunoprécipitation de chromatine, ou ChIP-seq, combine la ChIP avec le séquençage de nouvelle génération (Barski 2007, Johnson 2007). Les protocoles ChIP-seq ont été adaptés des méthodes ChIP-chip : les protéines sont réticulées à leur ADN lié par un traitement au formaldéhyde, les cellules sont homogénéisées, et la chromatine est cisaillée et immunoprécipitée avec des billes magnétiques liées à des anticorps. L’ADN immunoprécipité est ensuite utilisé comme entrée pour un protocole de préparation de bibliothèque de séquençage de nouvelle génération, où il est séquencé et analysé pour les sites de liaison à l’ADN. Voir la figure ci-dessous pour un résumé du flux de travail ChIP-seq, et un exemple de résultats ChIP-seq (reproduit avec l’aimable autorisation de Dominic Schmidt (Schmidt 2009)).
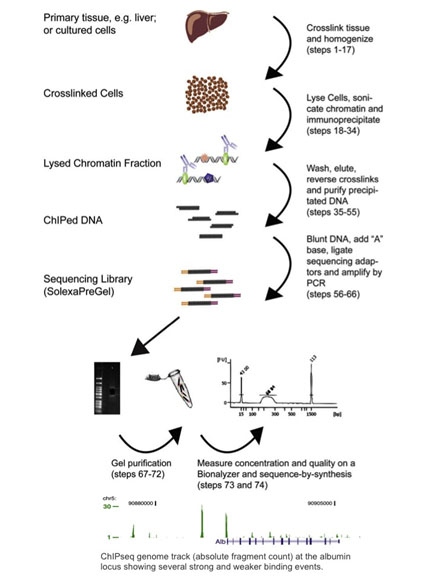
Bien que la majorité des quelque 400 articles publiés jusqu’à présent aient été analysés sur la plateforme Illumina, la ChIP-seq peut être réalisée sur n’importe quel séquenceur de nouvelle génération (Wold 2008). La technique ChIP-seq a été largement adoptée depuis qu’elle a été signalée pour la première fois en 2007. En fait, il a presque totalement supplanté le ChIP-Chip, car il permet une analyse à l’échelle du génome et n’a pas les limitations discutées ci-dessus.
Que pouvez-vous réellement faire avec ChIP-seq
ChIP-seq est un outil puissant et polyvalent, et il y a beaucoup de grands exemples d’utilisation de ChIP-seq dans la littérature. J’en ai choisi quelques-uns parmi mes préférés (issus de travaux réalisés dans l’installation centrale que je gère) pour illustrer ce qui est possible, et j’ai inclus des exemples où ChIP-seq a inspiré le développement de nouvelles méthodes :
- Antoni Hurtado, et al. ont réalisé un knock-down du “facteur pionnier” FoxA1, ce qui a entraîné une réduction de la liaison par le récepteur des œstrogènes (ER) à plus de 50% des sites de liaison ER connus. Ils ont montré que FoxA1 est un régulateur important de la transcription médiée par le RE, suggérant qu’il pourrait être une nouvelle cible thérapeutique importante dans le cancer du sein (Hurtado 2011).
- Dominic Shmidt, et al. ont utilisé ChIP-seq pour étudier l’évolution de la liaison des facteurs de transcription. Ils se sont concentrés sur la liaison de CEBPA et HNF4 dans le tissu hépatique de cinq espèces de vertébrés : l’homme, la souris, le chien, l’opossum et le poulet. La ChIP-chip aurait été presque impossible compte tenu des différentes espèces concernées et des complexités de conception des sondes (Schmidt 2010).
Des modifications ultérieures de la méthode ChIP-seq ont conduit à plusieurs nouvelles méthodes, notamment la ChIP-seq pour l’analyse des interactions ARN-protéines, ainsi que la DNase-seq et la FAIRE-seq, toutes deux utilisées pour identifier les régions régulatrices dans l’ADN.
En résumé, la ChIP-seq est une technique mature qui est préférable à la ChIP-chip et peut être utilisée par presque tous les groupes intéressés par l’analyse des interactions ADN:protéines. Comment pourriez-vous utiliser ChIP-seq dans votre recherche?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 est un déterminant clé de la fonction du récepteur des œstrogènes et de la réponse endocrinienne.Nature Genetics (2011).
Johnson et al, Cartographie à l’échelle du génome des interactions protéine-ADN in vivo. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Applications des technologies de séquençage de nouvelle génération en génomique fonctionnelle. Genomics (2008).
Schmidt et al, ChIP-seq : utilisation du séquençage à haut débit pour découvrir les interactions protéine-ADN. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Cela vous a aidé ? Alors partagez avec votre réseau.
.