A ChIP-seq egy csodálatos technika, amely lehetővé teszi a fehérje és a DNS közötti fizikai kötési kölcsönhatások vizsgálatát a következő generációs szekvenálás segítségével. Ebben a cikkben röviden áttekintem a ChIP-et, és bemutatom a kromatin immunprecipitációs szekvenálási technikát (ChIP-seq), amely kombinálja a ChIP-et az újgenerációs szekvenálással.
Mi az a kromatin immunprecipitáció?
A kromatin immunprecipitáció (ChIP) lehetővé teszi számunkra, hogy meghatározzuk a DNS-en lévő fehérje-kötőhelyeket. A kromatin a DNS-nek a hisztonfehérjékkel nukleoszómákba csomagolt komplexe. A ChIP a sejtek vagy szövetek formaldehid fixálásával a DNS és a kapcsolódó fehérjék között létrejött reverzibilis keresztkötéseket használja fel. A rögzített kromatint fizikailag megnyírják, és egy adott fehérjéhez kapcsolódó DNS-töredékeket szelektíven immunprecipitálnak és elemeznek. Az elemzés történhet lókuszonként PCR segítségével, de a ChIP-et leggyakrabban mikrotérképpel (ChIP-chip) vagy újgenerációs szekvenálással (ChIP-seq) vizsgálják.
Hogyan működik a ChIP-chip?
A ChIP-on-chip vagy ChIP-chip a kromatin immunprecipitációt és a mikrotérképpel (chip) végzett elemzést kombinálja. 2001-ben Jason Lieb, Pat Brown stanfordi laboratóriumában Jason Brown publikálta az első ChIP-chip-publikációt (Lieb 2001). Ennél a módszernél az adott fehérjével kicsapódó DNS-töredékeket egy microarray chipre viszik fel elemzésre. Ezáltal globális képet kapunk arról, hogy hol kötődik a fehérje, szemben az egyes kötőhelyek PCR-rel történő egyszerű lekérdezésével. Bár ez forradalmi megközelítés volt, a technikát korlátozzák az alkalmazott tömbtechnológiák. Először is, szükség van a vizsgálni kívánt genomhoz tartozó microarray-re, és a 2000-es évek elején és közepén ez gyakran nem volt így. Másodszor, ezeket a microarray-ket korlátozza a referencia genom minősége és az a képesség, hogy olyan szondákat tervezzenek, amelyek működnek egy array-n. Harmadszor, a tömbök korlátozottak abban, hogy a genom mekkora részét tudják reprezentálni, és általában a genomot 100 vagy 1000 bp-s felbontással fedik le. A ChIP DNS-fragmentumok amplifikációjának torzításával, a tömbi adatok normalizálásával és a tömbi platformok összehasonlíthatóságával kapcsolatos kérdések is felmerülnek.
Hogyan működik a ChIP-seq?
A chromatin immunprecipitációs szekvenálás vagy ChIP-seq a ChIP-et és az újgenerációs szekvenálást kombinálja (Barski 2007, Johnson 2007). A ChIP-seq protokollokat a ChIP-chip módszerekből adaptálták: a fehérjéket formaldehidkezeléssel keresztkötik a hozzájuk kötött DNS-hez, a sejteket homogenizálják, majd a kromatint nyírják és ellenanyaghoz kötött mágneses gyöngyökkel immunprecipitálják. Az immunprecipitált DNS-t ezután egy újgenerációs szekvenálási könyvtárkészítési protokoll bemeneteként használják, ahol szekvenálják és elemzik a DNS-kötőhelyeket. Az alábbi ábrán látható a ChIP-seq munkafolyamat összefoglalása, valamint egy példa a ChIP-seq eredményeire (Dominic Schmidt (Schmidt 2009) szíves engedélyével).
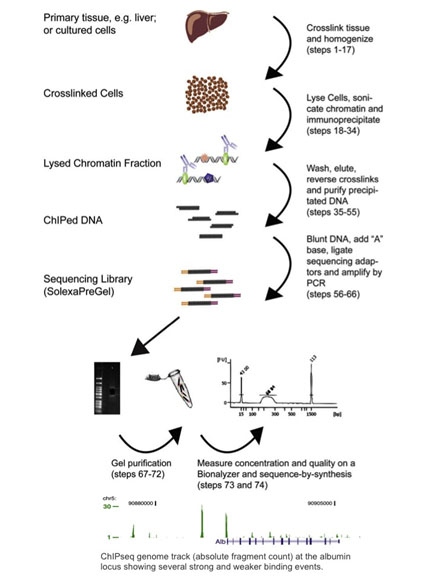
Noha az eddig megjelent mintegy 400 publikáció többségét az Illumina platformon elemezték, a ChIP-seq bármelyik újgenerációs szekvenálóval elvégezhető (Wold 2008). A ChIP-seq széles körben elterjedt, mióta 2007-ben először beszámoltak róla. Valójában szinte teljesen kiszorította a ChIP-Chipet, mivel lehetővé teszi az egész genomra kiterjedő elemzést, és nem rendelkezik a fent tárgyalt korlátozásokkal.
Mit lehet valójában csinálni a ChIP-seq-vel
A ChIP-seq hatékony és sokoldalú eszköz, és az irodalomban számos nagyszerű példát találunk a ChIP-seq alkalmazására. Kiválasztottam néhányat a kedvenceim közül (az általam vezetett core facility-ben végzett munkákból), hogy bemutassam, mi minden lehetséges, és olyan példákat is mellékeltem, ahol a ChIP-seq új módszerek kifejlesztését inspirálta:
- Antoni Hurtado, et al. a FoxA1 “úttörő faktor” knock-downját végezte el, ami az ösztrogénreceptor (ER) csökkent kötődését eredményezte az ismert ER-kötőhelyek több mint 50%-ánál. Kimutatták, hogy a FoxA1 az ER által közvetített transzkripció fontos szabályozója, ami arra utal, hogy új és fontos terápiás célpont lehet az emlőrákban (Hurtado 2011).
- Dominic Shmidt, és munkatársai ChIP-seq segítségével vizsgálták a transzkripciós faktorok kötődésének evolúcióját. Öt gerinces faj: ember, egér, kutya, oposszum és csirke májszövetében a CEBPA és a HNF4 kötődésére összpontosítottak. A ChIP-chip szinte lehetetlen lett volna, tekintettel az érintett fajok különbözőségére és a szondák tervezésének bonyolultságára (Schmidt 2010).
A ChIP-seq módszer további módosításai számos új módszert eredményeztek, köztük a ChIP-seq-t az RNS-fehérje kölcsönhatások elemzésére, valamint a DNase-seq-t és a FAIRE-seq-t, amelyeket a DNS-ben található szabályozó régiók azonosítására használnak.
Összefoglalva, a ChIP-seq egy kiforrott technika, amely előnyösebb, mint a ChIP-chip, és szinte bármely, a DNS:fehérje kölcsönhatások elemzése iránt érdeklődő csoport használhatja. Hogyan használhatná a ChIP-seq-et a kutatásában?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Applications of next-generation sequencing technologies in functional genomics. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Has this helped you? Akkor kérjük, ossza meg a hálózatával.