ChIP-seq è una tecnica meravigliosa che ci permette di interrogare le interazioni fisiche di legame tra proteine e DNA utilizzando il sequenziamento di prossima generazione. In questo articolo, darò una breve rassegna di ChIP e introdurrò la tecnica di sequenziamento dell’immunoprecipitazione della cromatina (ChIP-seq), che combina ChIP con il sequenziamento di prossima generazione.
Cos’è l’immunoprecipitazione della cromatina?
L’immunoprecipitazione della cromatina (ChIP) ci permette di determinare i siti di legame delle proteine sul DNA. La cromatina è il complesso di DNA impacchettato con proteine istone in nucleosomi. La ChIP fa uso di legami incrociati reversibili creati tra il DNA e le proteine associate dalla fissazione in formaldeide di cellule o tessuti. La cromatina fissata viene fisicamente tagliata e i frammenti di DNA associati a una particolare proteina vengono selettivamente immunoprecipitati e analizzati. L’analisi può essere su base locus-by-locus utilizzando la PCR, ma più comunemente ChIP è interrogato con microarray (ChIP-chip) o sequenziamento di prossima generazione (ChIP-seq).
Come funziona ChIP-chip?
ChIP-on-chip, o ChIP-chip, combina l’immunoprecipitazione della cromatina con analisi microarray (chip). Nel 2001, Jason Lieb nel laboratorio di Pat Brown a Stanford ha pubblicato il primo articolo ChIP-chip (Lieb 2001). In questo metodo, i frammenti di DNA che precipitano con una particolare proteina sono applicati a un chip microarray per l’analisi. Questo genera un quadro globale di dove la proteina si lega, in contrasto con la semplice interrogazione di singoli siti di legame tramite PCR. Mentre questo è stato un approccio rivoluzionario, la tecnica è limitata dalle tecnologie di array utilizzate. In primo luogo, è necessario un microarray per il genoma che si vuole studiare, e nei primi anni 2000 questo spesso non era il caso. In secondo luogo, quei microarray sono limitati dalla qualità del genoma di riferimento e dalla capacità di progettare sonde che funzionino su un array. In terzo luogo, gli array sono limitati nella misura in cui il genoma può essere rappresentato, e di solito si sovrappongono attraverso il genoma a 100 o 1000 bp di risoluzione. Ci sono anche problemi di bias nell’amplificazione dei frammenti di DNA ChIP, la normalizzazione dei dati di array, e la comparabilità delle piattaforme di array.
Come funziona il ChIP-seq?
Il sequenziamento di immunoprecipitazione della cromatina, o ChIP-seq, combina ChIP con il sequenziamento di prossima generazione (Barski 2007, Johnson 2007). I protocolli ChIP-seq sono stati adattati dai metodi ChIP-chip: le proteine sono reticolate al loro DNA legato dal trattamento con formaldeide, le cellule sono omogeneizzate, e la cromatina viene tosata e immunoprecipitata con anticorpi legati a perline magnetiche. Il DNA immunoprecipitato viene quindi utilizzato come input per un protocollo di preparazione della libreria di sequenziamento di prossima generazione, dove viene sequenziato e analizzato per i siti di legame al DNA. Vedi la figura qui sotto per una sintesi del flusso di lavoro ChIP-seq, e un esempio di risultati ChIP-seq (riprodotto con il permesso di Dominic Schmidt (Schmidt 2009)).
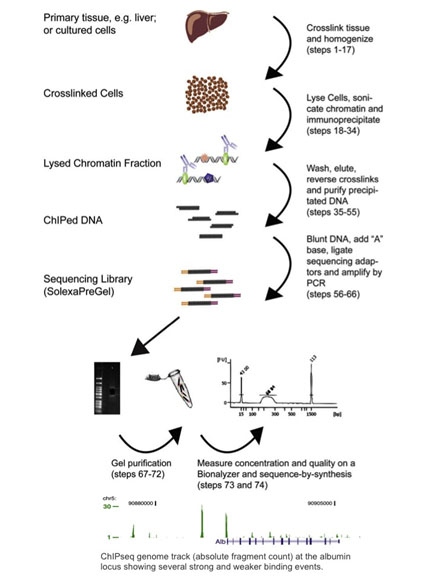
Anche se la maggior parte dei circa 400 articoli pubblicati finora sono stati analizzati sulla piattaforma Illumina, ChIP-seq può essere eseguita su qualsiasi sequenziatore di prossima generazione (Wold 2008). ChIP-seq è stato ampiamente adottato da quando è stato riportato per la prima volta nel 2007. Infatti, ha quasi totalmente soppiantato ChIP-Chip, dal momento che permette l’analisi genome-wide e non ha le limitazioni discusse in precedenza.
Cosa si può effettivamente fare con ChIP-seq
ChIP-seq è uno strumento potente e versatile, e ci sono molti grandi esempi di utilizzo ChIP-seq in letteratura. Ho scelto un paio dei miei preferiti (dal lavoro svolto nel centro che gestisco) per illustrare ciò che è possibile, e ho incluso esempi in cui ChIP-seq ha ispirato lo sviluppo di nuovi metodi:
- Antoni Hurtado, et al. hanno eseguito il knock-down del “fattore pioniere” FoxA1, con conseguente riduzione del legame del recettore degli estrogeni (ER) in oltre il 50% dei siti di legame ER conosciuti. Hanno dimostrato che FoxA1 è un importante regolatore della trascrizione ER-mediata, suggerendo che potrebbe essere un nuovo e importante bersaglio terapeutico nel cancro al seno (Hurtado 2011).
- Dominic Shmidt, et al. hanno usato ChIP-seq per studiare l’evoluzione del legame dei fattori di trascrizione. Si sono concentrati sul legame di CEBPA e HNF4 nel tessuto epatico di cinque specie di vertebrati: uomo, topo, cane, opossum e pollo. ChIP-chip sarebbe stato quasi impossibile, date le diverse specie coinvolte e le complessità nella progettazione delle sonde (Schmidt 2010).
Altre modifiche al metodo ChIP-seq hanno portato a diversi nuovi metodi, tra cui ChIP-seq per l’analisi dell’interazione RNA-proteina, così come DNase-seq e FAIRE-seq, entrambi utilizzati per identificare le regioni di regolazione nel DNA.
In sintesi, ChIP-seq è una tecnica matura che è preferibile a ChIP-chip e può essere utilizzata da quasi tutti i gruppi con un interesse nell’analisi di interazione DNA:Proteina. Come potresti usare ChIP-seq nella tua ricerca?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 è un determinante chiave della funzione del recettore degli estrogeni e della risposta endocrina.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Scienza (2007).
Lee et al, Cromatina immunoprecipitazione e microarray-based analisi della posizione della proteina. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Applicazioni delle tecnologie di sequenziamento di prossima generazione nella genomica funzionale. Genomics (2008).
Schmidt et al, ChIP-seq: utilizzando il sequenziamento ad alta velocità per scoprire le interazioni proteina-DNA. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Ti è stato utile? Allora per favore condividi con la tua rete.