ChIP-seq は、次世代シーケンサーを用いて、タンパク質とDNAの物理的な結合相互作用を調べることができる素晴らしい技術です。 今回は、ChIPの簡単なおさらいと、ChIPと次世代シーケンサーを組み合わせたクロマチン免疫沈降シーケンス技術(ChIP-seq)について紹介します。
クロマチン免疫沈降とは
ChIP (Chromatin immunoprecipitation) によりDNA上のタンパク質結合部位が決定されることが知られています。 クロマチンとは、DNAがヒストン蛋白質とともにヌクレオソームに包まれた複合体のことです。 ChIPは、細胞や組織をホルムアルデヒドで固定することにより、DNAと関連タンパク質が可逆的に架橋されることを利用する。 固定されたクロマチンを物理的に切断し、特定のタンパク質に結合したDNA断片を選択的に免疫沈降させ、分析する。 PCRを用いた遺伝子座ごとの解析も可能ですが、より一般的にはマイクロアレイ(ChIP-chip)または次世代シーケンサー(ChIP-seq)を用いてChIPを解析します。 2001年にスタンフォード大学のPat Brownの研究室のJason Liebが最初のChIP-chipの論文を発表しました(Lieb 2001)。 この方法では、特定のタンパク質と沈殿したDNA断片を、マイクロアレイチップに載せて解析する。 この方法では、単にPCRで1つの結合部位を調べるのとは対照的に、タンパク質が結合する場所の全体像が明らかになる。 これは画期的なアプローチだが、使用するアレイ技術によって、この手法には限界がある。 まず、研究したいゲノムのマイクロアレイが必要ですが、2000年代前半から半ばにかけては、そうでないことがよくありました。 次に、それらのマイクロアレイは、参照ゲノムの品質と、アレイ上で機能するプローブを設計する能力によって制限される。 第三に、アレイはゲノムのどの部分を表現できるかという点で限界があり、通常、100から1000bpの分解能でゲノム全体にタイル状に配置されている。 また、ChIP DNAフラグメントの増幅におけるバイアス、アレイデータの正規化、アレイプラットフォームの比較可能性についても問題があります。 ChIP-seqプロトコルは、ChIP-chip法から転用されたもので、タンパク質はホルムアルデヒド処理によって結合DNAに架橋され、細胞はホモジナイズされ、クロマチンはせん断されて抗体結合磁気ビーズで免疫沈降される。 免疫沈降したDNAは、次世代シーケンサーのライブラリープレッププロトコルのインプットとして使用され、DNA結合部位の配列決定と解析が行われる。 ChIP-seqワークフローの概要とChIP-seq結果の例については、以下の図を参照してください(Dominic Schmidt (Schmidt 2009)の許可を得て複製)
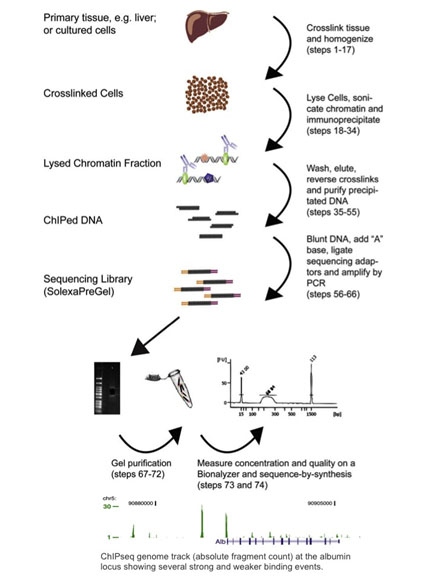
これまでに発表された400ほどの論文の大半は、イルミナのプラットフォームで分析されていますが、ChIP-seqはあらゆる次世代のシーケンサーで行うことができます(Wold 2008)。 ChIP-seqは、2007年に初めて報告されて以来、広く採用されるようになりました。 実際、ゲノムワイドな解析が可能で、上で述べたような制限もないため、ほぼ完全にChIP-Chipに取って代わりました。
ChIP-seq で実際にできること
ChIP-seq は強力なツールで汎用性が高く、文献上にも多くの素晴らしい使用例が掲載されています。 Antoni Hurtado氏らは、FoxA1「パイオニア因子」のノックダウンを行い、既知のER結合部位の50%以上でエストロゲン受容体(ER)による結合が減少する結果を得ました。 彼らはFoxA1がERを介した転写の重要な制御因子であることを示し、乳癌の新しい重要な治療ターゲットになる可能性を示唆した(Hurtado 2011)
ChIP-seq手法にさらに手を加えた結果、RNA-タンパク質の相互作用解析用のChIP-seq、DNAにおける制御領域の特定に用いられるDNase-seqおよびFAIREのseqなどの新しい手法が出ている。
まとめると、ChIP-seqはChIP-chipよりも望ましい成熟した技術であり、DNA:タンパク質相互作用解析に関心を持つほぼすべてのグループが使用することができるものです。
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions.「ゲノムにおけるタンパク質-DNA相互作用のマッピング」. また、このような研究成果を踏まえて、「ゲノムワイドな蛋白質-DNA相互作用のマッピング」、「クロマチン免疫沈降法とマイクロアレイを用いた蛋白質位置の解析」を行いました。 Nat. プロトコール (2006).
Lieb et al, Rap1のプロモーター特異的結合をゲノムワイドなタンパク質-DNA結合マップにより明らかにした。 Nat Genet. 2001.
Morozova & Marra, Applications of next-generation sequencing technologies in functional genomics.日本学術振興会特別研究員(PD)。 Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions.日本経済新聞社(2008).
Schmidt et al, Inc. また、このような研究成果をもとに、ゲノム解析のためのシーケンスセンサーの開発を進めています。 Nat. Methods (2008).
これはあなたのお役に立ちましたか?