ChIP-seq é uma técnica maravilhosa que nos permite interrogar as interacções de ligação física entre as proteínas e o ADN utilizando a sequenciação da próxima geração. Neste artigo, vou fazer uma breve revisão do ChIP e introduzir a técnica de imunoprecipitação por cromatina (ChIP-seq), que combina o ChIP com a sequência de próxima geração.
O que é imunoprecipitação por cromatina?
Imunoprecipitação por cromatina (ChIP) permite-nos determinar os sítios de ligação de proteínas no ADN. A cromatina é o complexo de ADN embalado com proteínas histone em nucleossomas. O ChIP faz uso de ligações cruzadas reversíveis feitas entre o DNA e as proteínas associadas através da fixação de formaldeído de células ou tecidos. A cromatina fixa é tosquiada fisicamente e os fragmentos de DNA associados a uma determinada proteína são seletivamente imunoprecipitados e analisados. A análise pode ser feita locus-by-locus usando PCR, mas mais comumente o ChIP é interrogado com microarrays (ChIP-chip) ou sequenciamento de próxima geração (ChIP-seq).
Como funciona o ChIP-chip?
ChIP-on-chip, ou ChIP-chip, combina imunoprecipitação de cromatina com análise de microarranjo (chip). Em 2001, Jason Lieb no laboratório de Pat Brown em Stanford publicou o primeiro artigo sobre ChIP-chip (Lieb 2001). Neste método, os fragmentos de DNA que precipitam com uma determinada proteína são aplicados a um chip de microarranjo para análise. Isto gera um quadro global de onde a proteína se liga, em contraste com a simples interrogação de locais de ligação únicos por PCR. Embora esta tenha sido uma abordagem revolucionária, a técnica é limitada pelas tecnologias de array utilizadas. Primeiramente, você precisa de um microarray para o genoma que você quer estudar, e no início a meados dos anos 2000 isso muitas vezes não era o caso. Em segundo lugar, esses microarrays são limitados pela qualidade do genoma de referência e pela capacidade de projetar sondas que funcionarão em um array. Em terceiro lugar, os arrays são limitados em quanto do genoma pode ser representado, e normalmente ladrilho através do genoma a 100’s ou 1000’s de resolução bp. Há também problemas sobre o viés na amplificação dos fragmentos de DNA ChIP, normalização dos dados de array e comparabilidade das plataformas de array.
Como o ChIP-seq funciona?
Sequenciamento de imunoprecipitação de cromatina, ou ChIP-seq, combina ChIP com sequenciamento de próxima geração (Barski 2007, Johnson 2007). Os protocolos ChIP-seq foram adaptados dos métodos ChIP-chip: as proteínas são reticuladas ao seu ADN ligado através do tratamento com formaldeído, as células são homogeneizadas e a cromatina é tosquiada e imunoprecipitada com contas magnéticas ligadas a anticorpos. O ADN imunoprecipitado é então utilizado como entrada para um protocolo de preparação da biblioteca de sequenciação da próxima geração, onde é sequenciado e analisado para locais de ligação do ADN. Veja a figura abaixo para um resumo do fluxo de trabalho ChIP-seq, e um exemplo dos resultados ChIP-seq (reproduzidos com a permissão de Dominic Schmidt (Schmidt 2009)).
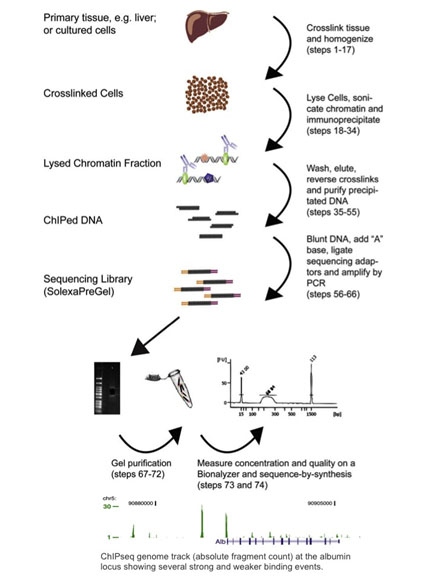
Embora a maioria dos cerca de 400 trabalhos publicados até agora tenham sido analisados na plataforma Illumina, o ChIP-seq pode ser realizado em qualquer sequenciador da próxima geração (Wold 2008). O ChIP-seq tem sido amplamente adotado desde que foi relatado pela primeira vez em 2007. Na verdade, ele tem quase totalmente suplantado o ChIP-Chip, uma vez que ele permite a análise de todo o genoma e não tem as limitações discutidas acima.
O que você pode realmente fazer com ChIP-seq
ChIP-seq é uma ferramenta poderosa e versátil, e há muitos grandes exemplos de uso do ChIP-seq na literatura. Escolhi alguns dos meus favoritos (de trabalhos realizados nas instalações centrais que administro) para ilustrar o que é possível, e incluí exemplos onde o ChIP-seq inspirou o desenvolvimento de novos métodos:
- Antoni Hurtado, et al. realizaram knock-down do “fator pioneiro” FoxA1, resultando na redução da ligação pelo receptor de estrogênio (ER) em mais de 50% dos locais de ligação do ER conhecidos. Eles mostraram que o FoxA1 é um importante regulador da transcrição mediada por ER, sugerindo que ele pode ser um novo e importante alvo terapêutico no câncer de mama (Hurtado 2011).
- Dominic Shmidt, et al. usaram o ChIP-seq para investigar a evolução da ligação do fator de transcrição. Eles focaram a ligação de CEBPA e HNF4 no tecido hepático de cinco espécies de vertebrados: humano, rato, cão, opossum e galinha. O ChIP-chip teria sido quase impossível dadas as diferentes espécies envolvidas e complexidades no desenho das sondas (Schmidt 2010).
Outras modificações ao método ChIP-seq levaram a vários novos métodos, incluindo o ChIP-seq para análise da interação RNA-proteína, assim como o DNase-seq e FAIRE-seq, ambos utilizados para identificar regiões reguladoras no DNA.
Em resumo, ChIP-seq é uma técnica madura que é preferível ao ChIP-chip e pode ser usada por quase qualquer grupo com interesse na análise da interação DNA:Protein interaction analysis. Como você poderia usar o ChIP-seq em sua pesquisa?
Barski et al, perfil de alta resolução de metilações de histone no genoma humano. Cell 129 (2007).
Hurtado et al, FOXA1 é um determinante chave da função do receptor de estrogênio e resposta endócrina. Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Aplicações de tecnologias de sequenciamento de próxima geração em genômica funcional. Genomics (2008).
Schmidt et al, ChIP-seq: Usando seqüenciamento de alto rendimento para descobrir interações proteína-ADN. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Revele the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Ajudou-lhe isto? Então por favor partilhe com a sua rede.