ChIP-seq este o tehnică minunată care ne permite să interogăm interacțiunile fizice de legare dintre proteine și ADN utilizând secvențierea de generație următoare. În acest articol, voi face o scurtă trecere în revistă a ChIP și voi introduce tehnica de secvențiere a imunoprecipitării cromatinei (ChIP-seq), care combină ChIP cu secvențierea de generație următoare.
Ce este imunoprecipitarea cromatinei?
Imunoprecipitarea cromatinei (ChIP) ne permite să determinăm situsurile de legare a proteinelor pe ADN. Cromatina este complexul de ADN împachetat cu proteine histone în nucleozomi. ChIP se folosește de legăturile încrucișate reversibile realizate între ADN și proteinele asociate prin fixarea cu formaldehidă a celulelor sau a țesuturilor. Cromatina fixată este forfecată fizic, iar fragmentele de ADN asociate cu o anumită proteină sunt imunoprecipitate și analizate în mod selectiv. Analiza poate fi efectuată locus cu locus cu ajutorul PCR, dar, mai frecvent, ChIP este interogată cu ajutorul microrețelelor (ChIP-chip) sau al secvențierii de generație următoare (ChIP-seq).
Cum funcționează ChIP-chip?
ChIP-on-chip, sau ChIP-chip, combină imunoprecipitarea cromatinei cu analiza microrețelelor (cip). În 2001, Jason Lieb din laboratorul lui Pat Brown de la Stanford a publicat prima lucrare ChIP-chip (Lieb 2001). În această metodă, fragmentele de ADN care precipită cu o anumită proteină sunt aplicate pe un cip microarray pentru analiză. Acest lucru generează o imagine globală a locului în care proteina se leagă, spre deosebire de simpla interogare a situsurilor unice de legare prin PCR. Deși aceasta a fost o abordare revoluționară, tehnica este limitată de tehnologiile de matrice utilizate. În primul rând, aveți nevoie de un microarray pentru genomul pe care doriți să îl studiați, iar la începutul sau la mijlocul anilor 2000 acest lucru nu era adesea cazul. În al doilea rând, aceste microrețele sunt limitate de calitatea genomului de referință și de capacitatea de a proiecta sonde care să funcționeze pe o matrice. În al treilea rând, microrețelele sunt limitate în ceea ce privește partea din genom care poate fi reprezentată și, de obicei, acoperă tot genomul la o rezoluție de 100 sau 1000 de bp. Există, de asemenea, probleme legate de distorsiunea în amplificarea fragmentelor de ADN ChIP, normalizarea datelor de matrice și comparabilitatea platformelor de matrice.
Cum funcționează ChIP-seq?
Secvențierea prin imunoprecipitare cu cromatină, sau ChIP-seq, combină ChIP cu secvențierea de generație următoare (Barski 2007, Johnson 2007). Protocoalele ChIP-seq au fost adaptate din metodele ChIP-chip: proteinele sunt reticulate la ADN-ul lor legat prin tratament cu formaldehidă, celulele sunt omogenizate, iar cromatina este forfecată și imunoprecipitată cu margele magnetice legate de anticorpi. ADN-ul imunoprecipitat este apoi utilizat ca intrare pentru un protocol de pregătire a unei biblioteci de secvențiere de generație următoare, unde este secvențiat și analizat pentru situsurile de legare a ADN-ului. A se vedea figura de mai jos pentru un rezumat al fluxului de lucru ChIP-seq și un exemplu de rezultate ChIP-seq (reprodus cu permisiunea amabilă a lui Dominic Schmidt (Schmidt 2009)).
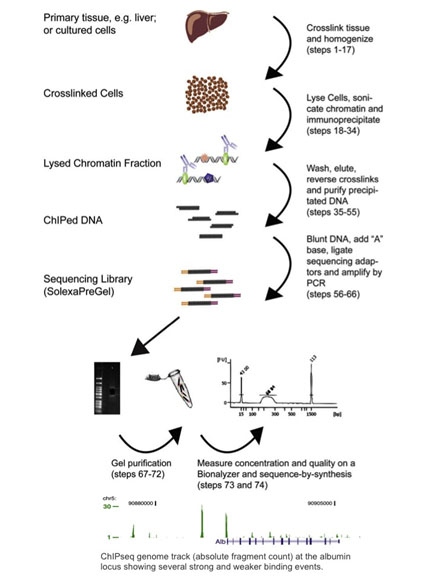
Deși majoritatea celor aproximativ 400 de lucrări publicate până în prezent au fost analizate pe platforma Illumina, ChIP-seq poate fi efectuată pe orice secvențiator de generație următoare (Wold 2008). ChIP-seq a fost adoptat pe scară largă de când a fost raportat pentru prima dată în 2007. De fapt, a suplinit aproape în totalitate ChIP-Chip, deoarece permite o analiză la nivelul întregului genom și nu are limitările discutate mai sus.
Ce se poate face de fapt cu ChIP-seq
ChIP-seq este un instrument puternic și versatil, iar în literatura de specialitate există multe exemple excelente de utilizare a ChIP-seq. Am ales câteva dintre preferatele mele (din lucrările efectuate în cadrul instalației de bază pe care o administrez) pentru a ilustra ceea ce este posibil și am inclus exemple în care ChIP-seq a inspirat dezvoltarea de noi metode:
- Antoni Hurtado, et al. au efectuat knock-down al “factorului pionier” FoxA1, ceea ce a dus la reducerea legării de către receptorul de estrogen (ER) la peste 50% din situsurile cunoscute de legare a ER. Ei au arătat că FoxA1 este un regulator important al transcripției mediate de ER, sugerând că ar putea fi o țintă terapeutică nouă și importantă în cancerul de sân (Hurtado 2011).
- Dominic Shmidt, et al. au folosit ChIP-seq pentru a investiga evoluția legării factorilor de transcripție. Ei s-au concentrat pe legarea CEBPA și HNF4 în țesutul hepatic a cinci specii de vertebrate: om, șoarece, câine, oposum și pui. ChIP-chip ar fi fost aproape imposibil, având în vedere diferitele specii implicate și complexitatea conceperii sondelor (Schmidt 2010).
Modificări ulterioare ale metodei ChIP-seq au condus la mai multe metode noi, inclusiv ChIP-seq pentru analiza interacțiunii ARN-proteine, precum și DNase-seq și FAIRE-seq, ambele utilizate pentru a identifica regiunile de reglementare din ADN.
În concluzie, ChIP-seq este o tehnică matură care este preferabilă lui ChIP-chip și poate fi utilizată de aproape orice grup interesat de analiza interacțiunii ADN:proteină. Cum ați putea utiliza ChIP-seq în cercetarea dumneavoastră?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Aplicații ale tehnologiilor de secvențiere de generație următoare în genomica funcțională. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
V-a ajutat acest lucru? Atunci vă rugăm să împărtășiți cu rețeaua dumneavoastră.
.