ChIP-seq er en fantastisk teknik, der giver os mulighed for at undersøge de fysiske bindingsinteraktioner mellem protein og DNA ved hjælp af næste generations sekventering. I denne artikel giver jeg en kort gennemgang af ChIP og introducerer kromatinimmunoprecipitations-sekventeringsteknikken (ChIP-seq), som kombinerer ChIP med næste generations sekventering.
Hvad er kromatinimmunoprecipitation?
Kromatinimmunoprecipitation (ChIP) giver os mulighed for at bestemme proteinbindingssteder på DNA. Kromatin er det kompleks af DNA, der er pakket sammen med histonproteiner i nukleosomer. ChIP gør brug af reversible krydsforbindelser mellem DNA og associerede proteiner ved formaldehydfiksering af celler eller væv. Det fikserede kromatin skæres fysisk, og DNA-fragmenter, der er associeret med et bestemt protein, immunudfældes selektivt og analyseres. Analysen kan foretages locus for locus ved hjælp af PCR, men oftest undersøges ChIP med mikroarrays (ChIP-chip) eller næste generations sekventering (ChIP-seq).
Hvordan fungerer ChIP-chip?
ChIP-on-chip, eller ChIP-chip, kombinerer kromatinimmunopræcipitation med mikroarray (chip)-analyse. I 2001 offentliggjorde Jason Lieb i Pat Browns laboratorium på Stanford den første ChIP-chip-artikel i 2001 (Lieb 2001). Ved denne metode påføres de DNA-fragmenter, der udfældes med et bestemt protein, på en mikroarray-chip til analyse. Dette giver et globalt billede af, hvor proteinet binder sig, i modsætning til blot at udspørge enkelte bindingssteder ved hjælp af PCR. Selv om dette var en revolutionerende tilgang, er teknikken begrænset af de anvendte array-teknologier. For det første skal man have et mikroarray til det genom, man ønsker at undersøge, og i begyndelsen og midten af 2000’erne var dette ofte ikke tilfældet. For det andet er disse mikroarrays begrænset af kvaliteten af referencegenomet og evnen til at designe prober, der kan fungere på et array. For det tredje er arrays begrænset med hensyn til, hvor stor en del af genomet der kan repræsenteres, og de er som regel placeret på tværs af genomet med en opløsning på 100 eller 1000 bp. Der er også problemer med bias i forbindelse med forstærkning af ChIP-DNA-fragmenterne, normalisering af array-data og sammenlignelighed af array-platforme.
Hvordan fungerer ChIP-seq?
Chromatin immunoprecipitation sequencing, eller ChIP-seq, kombinerer ChIP med næste generations sekventering (Barski 2007, Johnson 2007). ChIP-seq-protokoller er blevet tilpasset fra ChIP-chip-metoder: proteiner krydsbindes til deres bundne DNA ved hjælp af formaldehydbehandling, celler homogeniseres, og kromatin skæres og immunopræcipiteres med antistofbundne magnetiske perler. Det immunoprecipiterede DNA anvendes derefter som input til en protokol til forberedelse af et næste generations sekventeringsbibliotek, hvor det sekventeres og analyseres for DNA-bindingssteder. Se figuren nedenfor for en oversigt over ChIP-seq-arbejdsgangen og et eksempel på ChIP-seq-resultater (gengivet med venlig tilladelse fra Dominic Schmidt (Schmidt 2009)).
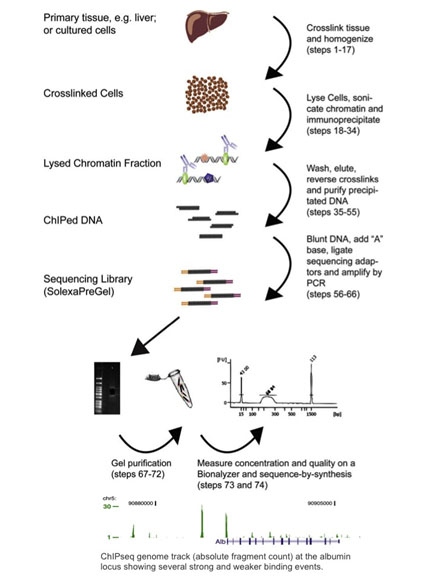
Og selv om størstedelen af de ca. 400 artikler, der er offentliggjort indtil nu, er blevet analyseret på Illumina-platformen, kan ChIP-seq udføres på enhver næste-generations-sequencer (Wold 2008). ChIP-seq er blevet bredt anvendt, siden det blev rapporteret første gang i 2007. Faktisk har det næsten helt fortrængt ChIP-Chip, da det giver mulighed for genomdækkende analyser og ikke har de begrænsninger, der er diskuteret ovenfor.
Hvad kan man egentlig gøre med ChIP-seq
ChIP-seq er et kraftfuldt og alsidigt værktøj, og der er mange gode eksempler på ChIP-seq-anvendelse i litteraturen. Jeg har udvalgt et par af mine favoritter (fra arbejde udført i den kernefacilitet, jeg leder) for at illustrere, hvad der er muligt, og har inkluderet eksempler, hvor ChIP-seq har inspireret til udvikling af nye metoder:
- Antoni Hurtado, et al. udførte knock-down af FoxA1 “pionerfaktoren”, hvilket resulterede i reduceret binding af østrogenreceptoren (ER) på over 50% af de kendte ER-bindingssteder. De viste, at FoxA1 er en vigtig regulator af ER-medieret transkription, hvilket tyder på, at den kan være et nyt og vigtigt terapeutisk mål i brystkræft (Hurtado 2011).
- Dominic Shmidt, et al. brugte ChIP-seq til at undersøge udviklingen af transkriptionsfaktorbinding. De fokuserede på CEBPA- og HNF4-binding i levervæv fra fem hvirveldyrsarter: menneske, mus, hund, hund, opossum og kylling. ChIP-chip ville have været næsten umuligt på grund af de forskellige arter, der var involveret, og kompleksiteten i udformningen af prober (Schmidt 2010).
Flere modifikationer af ChIP-seq-metoden har ført til flere nye metoder, herunder ChIP-seq til analyse af RNA-proteininteraktion samt DNase-seq og FAIRE-seq, som begge bruges til at identificere regulatoriske regioner i DNA.
Sammenfattende er ChIP-seq en moden teknik, der er at foretrække frem for ChIP-chip og kan anvendes af næsten alle grupper med interesse i DNA:Protein-interaktionsanalyse. Hvordan kan du bruge ChIP-seq i din forskning?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation og mikroarray-baseret analyse af proteinplacering. Nat. Protoc (2006).
Lieb et al, Promoter-specifik binding af Rap1 afsløret af genom-dækkende kort over protein-DNA-association. Nat Genet. 2001.
Morozova & Marra, Applications of next-generation sequencing technologies in functional genomics. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).

Har dette hjulpet dig? Så del venligst med dit netværk.