Vorwort
Dieses Papier bietet eine persönliche Vision dessen, was notwendig sein könnte, damit die Phagentherapie endlich den Durchbruch als allgemeines antibakterielles Mittel schafft. Er ist beeinflusst von historischen und aktuellen Misserfolgen und Unsicherheiten auf dem Gebiet der Phagentherapie und zielt darauf ab, Lösungen zu finden, die auf zukünftigen und aufkommenden Technologien basieren, die die Wissenschaft und Gesellschaft von morgen modellieren sollen.
Phagentherapie
Bakteriophagen (Phagen) sind die Viren der Bakterien. Seit Menschengedenken kontrollieren sie das Wachstum und die Ausbreitung ihrer bakteriellen Wirte. Bakterienviren sind die am weitesten verbreiteten Lebewesen in unserer Biosphäre. Es gibt schätzungsweise 10 Millionen Mal mehr Viren in den Ozeanen als Sterne im Universum, und wenn man alle Phagen auf der Erde übereinander stapeln würde, würde sich dieser Turm weiter erstrecken als die nächsten 60 Galaxien (Suttle, 2013). Sie sind überall dort zu finden, wo Bakterien gedeihen: in der Kanalisation, in Flüssen oder im Urin und Stuhl von Patienten. Phagen menschlicher bakterieller Krankheitserreger bestehen meist aus einem ikosaedrischen Kopf, einer Kugel mit 20 flachen Seiten, die aus Proteinen besteht und ein Nukleinsäuregenom enthält, an das ein Proteinschwanz angehängt ist. Wenn ein streng lytischer Phage mit seinen Schwanzfasern und Stacheln an der Oberfläche seines Zielbakteriums haftet, zieht sich die spritzenartige Schwanzhülle zusammen und der Schwanzkern wird durch die bakterielle Zellwand getrieben, wodurch das Phagengenom in das Periplasma der Bakterienzelle injiziert wird. Sofort wird die bakterielle DNA- und Proteinsynthesemaschinerie angezapft, um Kopien des Phagen zu erstellen. Einige Phagen zerschneiden die bakterielle DNA in Stücke. Nach einer Latenzzeit von Minuten bis Stunden brechen die neu gebildeten Phagen aus ihren bakteriellen Wirten aus, die dabei getötet werden. Die Nachkommen der Phagen, die in die Hunderte pro Bakterium gehen können, machen sich dann auf die Suche nach neuen Wirtsbakterien, um diese zu infizieren. Phagen können somit als selbstreplizierende antimikrobielle Mittel betrachtet werden. Wichtig ist, dass Phagen sich so entwickelt haben, dass sie nur bestimmte Zielbakterien infizieren und für Säugetierzellen unschädlich sind.
Frühe Hinweise auf virusähnliche Agenzien mit antibakterieller Wirkung wurden von dem englischen Bakteriologen Frederick Twort und dem französisch-kanadischen Mikrobiologen Felix d’Hérelle in den Jahren 1915 bzw. 1917 gemeldet (Sulakvelidze et al., 2001). 1919 nutzte d’Hérelle zum ersten Mal das therapeutische Potenzial von Phagen, als er sie zur Heilung eines an Dysenterie erkrankten Jungen in Paris einsetzte. Die Phagentherapie wurde sofort als therapeutischer Ansatz zur Behandlung bakterieller Infektionen anerkannt, und mehrere Unternehmen wie L’Oréal in Europa und Eli Lilly Company in den Vereinigten Staaten begannen mit der Vermarktung von Phagentherapie-Präparaten (Sulakvelidze et al., 2001). 1923 gründete der georgische Mikrobiologe Giorgi Eliava das Eliava-Institut in Tiflis, Georgien, das sich der Erforschung der Phagentherapie widmete. Dies war der Beginn umfangreicher Forschung und Entwicklung im Bereich der Phagentherapie in der ehemaligen Sowjetunion. Die ersten Anwendungen der Phagentherapie waren jedoch oft unzuverlässig, und auch die Forschung im Bereich der Antibiotika war bereits im Gange. Der erfolgreiche Einsatz von Penicillin während des Zweiten Weltkriegs und seine anschließende weltweite Vermarktung führten dazu, dass westliche Wissenschaftler das Interesse an der Phagentherapie verloren. Sowjetische Forscher hingegen entwickelten die Phagentherapie weiter und veröffentlichten ihre Ergebnisse, aber aufgrund des Eisernen Vorhangs verbreiteten sich ihre Kenntnisse und Erfahrungen nicht in der ganzen Welt (Sulakvelidze et al., 2001). Zu Beginn des dritten Jahrtausends weckte die zunehmende Gesundheitsbelastung durch Infektionen mit antibiotikaresistenten Bakterien (Cassini et al., 2019) ein erneutes weltweites Interesse an der Phagentherapie als praktikablem zusätzlichem Instrument für die klinische Behandlung bakterieller Infektionen (Thiel, 2004). Überall auf der Welt werden Phagentherapie-Zentren eingerichtet, die dem Beispiel des Eliava-Instituts und der Phagentherapie-Einheit am Hirszfeld-Institut in Wrocław, Polen, folgen (Miêdzybrodzki et al., 2012).
Das Jahr 2035
Wir schreiben das Jahr 2035, eine düstere Welt, die von menschlicher Überbevölkerung, großen Störungen des Ökosystems, globaler Erwärmung und Fremdenfeindlichkeit geprägt ist.
Während er in seiner Badewanne badete, verspürte Dr. John Iverian, ein Mikrobiologe im Ruhestand, plötzlich einen äußerst schmerzhaften Stich im Nacken, gefolgt von einem Geräusch, das an den Propeller eines kleinen Flugzeugs erinnerte. Er überprüfte die Umgebung und sah im Augenwinkel ein seltsames großes Insekt mit langen, unheimlichen Beinen und Antennen an der Wand neben seiner Designer-Badewanne sitzen. Osuri, das Hausverwaltungssystem von Iverians Loft im Zentrum von Antwerpen, identifizierte das Insekt als die braune marmorierte Stinkwanze Halyomorpha halys, die sich über die ganze Welt verbreitet hatte. In Osuris Bericht, der auf einen der Videobildschirme des Badezimmers projiziert wurde, hieß es, dass Menschen, die gebissen wurden, zunächst ein kleines rotes Geschwür an der Bissstelle ihrer Haut hatten. Wenn sie nicht behandelt wurden, schwoll die Bisswunde an und bildete Eiter. Müde und benebelt stieg Iverian aus dem Bad und ging ins Bett. Er hatte beschlossen, die aufwendige Wundbehandlung, zu der Osuri dringend geraten hatte, nicht durchzuführen. Am nächsten Morgen hatte sich die Bisswunde jedoch in eine nekrotische Wunde verwandelt, die deutliche Anzeichen einer Infektion aufwies.
Ängstlich aktivierte Iverian sein Phage-BEAM-Gerät. BEAM stand für “Bedside Energized Anti-Microbial”. Das Gerät hatte die Größe und Form eines Schuhkartons, aber mit einem eleganteren und polierten Aussehen. Der Name des Geräts und sein Hersteller waren in bunten Buchstaben auf der Seite der nahtlos weiß emaillierten Schachtel angebracht. Iverian nahm einen Tupfer aus der sterilen Verpackung und fuhr damit vorsichtig über den gesamten Bereich der Wunde, wobei er darauf achtete, dass das Wundexsudat die Wattespitze des Tupfers gut benetzte. Als sich der Tupfer dem Bereich “Probe einlegen” der Schachtel näherte, öffnete sich wie von Geisterhand eine winzige Tür und gab ein zehn Zentimeter hohes Hologramm einer Labortechnikerin namens Marcia frei. Sie zeigte Iverian, wo er die Probe einführen musste. Marcia wurde entwickelt, um die Kunden durch das Testverfahren zu führen. “Für optimale Ergebnisse setzen Sie bitte eine neue Phagen-Biotintenpatrone ein, Dr. Iverian”, sagte Marcia. Wie bei den 2D-Druckern vergangener Zeiten waren die Kosten für die Biotintenpatronen fast so hoch wie die Kosten für das Phage-BEAM-Gerät selbst. Laut “Business Insider” war Phagen-Biotinte die zweitteuerste Flüssigkeit der Welt, nach Chanel No. 8. Glücklicherweise hatte Iverian als einer der Erfinder das Recht erworben, immer die neueste Version dieses Geräts zur Verfügung zu haben, einschließlich eines ständigen Nachschubs an Reagenzien, und zwar kostenlos.
Iverian wusste genau, wie das Gerät funktionierte, so dass er Marcias Hilfe nicht benötigte. Zunächst wurde die DNA aus der Tupferspitze extrahiert und das Metagenom – das gesamte in der Probe vorhandene genetische Material, einschließlich der infizierenden Bakterien – bestimmt. Anschließend wurden diese genetischen Daten an einen gesicherten “Phage XChange”-Server gesendet, auf dem ein komplexer KI-gesteuerter Algorithmus die Genomsequenz des Phagen vorhersagte, der am ehesten in der Lage war, die im Metagenom identifizierten infizierenden Bakterien zu lysieren, und der beim Patienten die schwächste Immunreaktion hervorrufen sollte. Die Phagengenomdaten wurden an das Phage-BEAM-Gerät gesendet, das zunächst das Phagengenom und dann den Phagen mit Hilfe eines proprietären bakterienfreien Phagenproduktionssystems synthetisierte.
Innerhalb von 1 Stunde nach der Probenahme würde das Gerät ein gebrauchsfertiges therapeutisches Phagenprodukt herstellen. Die Ergebnisse des schrittweisen Verfahrens würden auf den riesigen Videobildschirm in Iverians Wohnzimmer übertragen. Iverian saß in seinem LC2-Sessel, hörte Mozarts Große Messe in c-Moll und wartete gespannt auf die Ergebnisse. Er hatte ein schlechtes Gefühl bei der Sache. Das Ergebnis ließ Iverian einen Schauer über den Rücken laufen. Bakterieller Erreger identifiziert: Streptococcus pyogenes Stamm FE-2033! Osuri aktivierte sofort das Infektionsalarm-Protokoll, schickte eine Nachricht an das World Center for Disease Control und lieferte beunruhigende Hintergrundinformationen über den tödlichen fleischfressenden Bakterienstamm, der seit 2033 als unmittelbare Bedrohung für die öffentliche Gesundheit galt. Einen Moment lang erwog Iverian, die infizierte Wunde und das umliegende gesunde Gewebe mit einem Küchenmesser herauszuschneiden, aber er beruhigte sich und beschloss, zu warten und das bevorstehende Phage-BEAM-Produkt anzuwenden. Eine Stunde später hatte das Phage-BEAM-Gerät synthetische Phagen hergestellt. Diese Phagen wurden dann in einem Validierungsmodul mit den isolierten Bakterien gemischt, um ihre In-vitro-Wirksamkeit zu testen. Fünfzehn Minuten später gab es grünes Licht für Iverian, mit der Behandlung zu beginnen. Iverian applizierte die Phagen in einem Hydrogel-Wundverband mit langsamer Freisetzung, der zuvor mit der vom Phage-BEAM-Gerät hergestellten konzentrierten Phagensuspension vermischt worden war und außerdem synergistische Antibiotika enthielt. Das Hydrogel linderte vorübergehend die Schmerzen, was ihn weiter beruhigte. Iverian wiederholte die Anwendung des mit Phagen und Antibiotika beladenen Hydrogels einmal pro Tag. Die Wundinfektion besserte sich innerhalb von 24 Stunden, und nach 7 Tagen war die Wunde fast vollständig abgeheilt. Iverians potenziell lebensbedrohliche Infektion wurde erfolgreich, rechtzeitig und ohne sein Haus zu verlassen, behandelt. Viele Jahrzehnte lang war jedoch nicht sicher, dass die Phagentherapie den Durchbruch zu einem breit angelegten und klinisch nützlichen antibakteriellen Mittel schaffen würde. Die medizinische Welt hatte eine Weile gebraucht, um zu erkennen, dass die Phagentherapie nicht mit der Antibiotikatherapie identisch sein musste, und dies vor allem wegen der Besonderheiten der Wirkstoffe, der Phagen.
Einige relevante Besonderheiten von Phagen
Phagen weisen eine Reihe von Eigenschaften auf, die sich von Antibiotika unterscheiden und ihre Entwicklung als Arzneimittel und ihre Anwendung in der Therapie behinderten. Erstens sind sie in der Regel sehr spezifisch in Bezug auf die Bakterien, die sie infizieren. Im besten Fall befallen sie einen beträchtlichen Teil einer einzigen Bakterienart, im schlimmsten Fall aber nur eine kleine Anzahl von Stämmen innerhalb einer Art. Therapeutische Phagen können daher so ausgewählt werden, dass sie hauptsächlich eine Bakterienart oder eine klinisch relevante Untergruppe davon abtöten und die nützlichen Bakterien des Patienten (z. B. die Darm-, Haut- oder orale Kommensalflora) verschonen. Die meisten routinemäßig eingesetzten Antibiotika haben dagegen ein breites Wirkungsspektrum, was zu “Kollateralschäden” an den kommensalen Mikrobiomen des Patienten führen kann, was wiederum unerwünschte Wirkungen wie die Selektion antibiotikaresistenter Bakterienarten (z. B. Clostridium difficile) oder Antibiotika-assoziierte Diarrhöe zur Folge haben kann (Jernberg et al., 2010). Der Nachteil der Phagenspezifität ist, dass die infizierenden Bakterien vor Beginn der Phagentherapie identifiziert werden müssen. Bei der empirischen Antibiotikatherapie werden dagegen in der Regel Breitspektrum-Antibiotika-Cocktails verwendet, die gegen eine Vielzahl von grampositiven und gramnegativen Bakterien sowie verschiedene Pilze wirken. Wenn mehr Informationen bekannt sind (z. B. aus Bakterienkulturen), kann die Behandlung aus Schmalbandantibiotika bestehen, die spezifischer auf die Bakterien oder Pilze abzielen, die als Verursacher der Krankheit identifiziert wurden.
Zweitens stehen Bakterien und Phagen in einer Wirt-Parasit-Beziehung. Streng lytische Phagen sind in der Umwelt allgegenwärtig und benötigen den Tod ihres bakteriellen Wirts, um ihren Lebenszyklus abzuschließen. Ohne Wirte können die Phagen nicht existieren. Phagen erzwingen eine Selektion auf resistente Wirte, die wiederum eine Selektion auf wirksame Phagen erzwingen. Daraus ergibt sich die so genannte “antagonistische Koevolution”, ein Wettrüsten zwischen Bakterien und Phagen, das durch eine wechselseitige Entwicklung von bakterieller Resistenz und Phageninfektiosität gekennzeichnet ist (Buckling und Rainey, 2002). Wie bei den meisten antimikrobiellen Mitteln werden Bakterien also auch gegen Phagen resistent (Luria und Delbrück, 1943; Schooley et al., 2017), aber im Gegensatz zu statischen Antibiotika haben Phagen die Fähigkeit, die bakterielle Resistenz zu überwinden (Buckling und Rainey, 2002). Es gibt jedoch Hinweise darauf, dass Bakterien und Phagen ihre jeweilige Resistenz und Infektiosität nicht unbegrenzt steigern werden (Fortuna et al., 2019).
Phagentherapieansätze
Zum Zeitpunkt des Phagentherapie-Revivals in den frühen 2000er Jahren wurden zwei verschiedene Phagentherapieansätze entwickelt (Pirnay et al., 2011). Beim so genannten “One-size-fits-all”-Ansatz wurden definierte Breitspektrum-Phagencocktails eingesetzt, die gegen die Mehrzahl der Bakterien wirken sollten, die im Verdacht stehen, bestimmte Infektionskrankheiten zu verursachen. Diese vordefinierten Breitspektrum-Phagencocktails wurden im Rahmen der derzeitigen pharmakoökonomischen Modelle entwickelt, hergestellt und getestet, die für “statische” Arzneimittel wie Antibiotika konzipiert worden waren. Echte Phagencocktails mit breitem Wirkungsspektrum, die gegen die meisten grampositiven und/oder gramnegativen Bakterien wirken, die bei Infektionskrankheiten häufig vorkommen, mussten jedoch große Mengen an Phagen enthalten, was sich als sehr schwierig erwies. Es war möglich, Phagencocktails mit einem engeren Spektrum zu entwickeln, die gegen eine oder wenige Bakterienarten wirksam sind und für bestimmte Indikationen eingesetzt werden können, sofern die infizierenden Bakterienarten im Voraus bekannt sind. Für einige Bakterienarten, wie z. B. Staphylococcus aureus, wurden Phagen mit einem außergewöhnlich breiten Wirtsspektrum isoliert und charakterisiert (Vandersteegen et al., 2011). In PhagoBurn, einer randomisierten kontrollierten Studie, war der Erfolg bei der nachhaltigen Verringerung der Belastung durch Pseudomonas aeruginosa in Verbrennungswunden mit der anfänglichen Empfänglichkeit für den Phagencocktail verbunden (Jault et al., 2019). Es zeigte sich jedoch, dass ein Drittel der eingeschlossenen Patienten bereits vorhandene P. aeruginosa-Stämme aufwies, die gegen den Phagencocktail resistent waren, der aus nicht weniger als 12 lytischen P. aeruginosa-Phagen bestand. Darüber hinaus mussten Phagencocktails, die ursprünglich als wirksam konzipiert waren, regelmäßig aktualisiert (z. B. durch neue Phagen ergänzt) werden, wenn eine Phagenresistenz auftrat oder neue klinisch relevante Stämme in Umlauf kamen. Schließlich war nicht bekannt, ob die Konfrontation von Bakterien mit hohen Konzentrationen fixierter Phagencocktails zur Entstehung, Verbreitung und Persistenz bakterieller Phagenresistenzen in Krankenhäusern und in der Umwelt führen würde, ähnlich wie es beim massiven Einsatz von Antibiotika geschehen war.
In personalisierten Phagentherapiekonzepten wurden ein oder mehrere Phagen aus einer Phagenbank oder aus der Umwelt ausgewählt und möglicherweise angepasst (In-vitro-Selektion von Phagenmutanten mit erhöhter Infektiosität), um die vom Infektionsort des Patienten isolierten Bakterien effizienter zu infizieren (Friman et al., 2016). Einige Phagentherapiezentren haben große therapeutische Phagenbanken eingerichtet und unterhalten, die regelmäßig mit neuen Phagen aktualisiert werden, um das Wirtsspektrum der Bank zu erweitern und an die sich ständig verändernden Bakterienpopulationen anzupassen. Personalisierte Phagentherapieansätze waren potenziell nachhaltiger, da nur das infizierende Bakterium angegriffen wird, was zu einem geringeren Selektionsdruck in Richtung bakterieller Phagenresistenz führt. Allerdings waren sie auch aufwändiger und logistisch komplexer als Einheitslösungen, da Bakterienstämme und passende Phagen in die ganze Welt verschickt werden mussten (Abbildung 1). Darüber hinaus waren die Konzepte der Präzisionsmedizin im Allgemeinen nicht mit den meisten Entwicklungs- und Zulassungsverfahren für Arzneimittel (in den USA) vereinbar, die mehrere Jahre und Millionen von Euro (Dollar) in Anspruch nahmen, und das für jeden Phagen in der Bank (Verbeken et al., 2012).
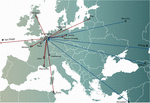
Abbildung 1. Internationale Transfers von Phagen aus (rote Pfeile) und in (blaue Pfeile) das Königin-Astrid-Militärkrankenhaus (QAMH) in Brüssel im Hinblick auf klinische Anwendungen im Zeitraum 2015-2020. Auf nationaler Ebene wurden Phagen vom QAMH an fünf Universitätskliniken versandt (nicht abgebildet). Darüber hinaus umfasste die Auswahl passender Phagen häufig die Übermittlung der bakteriellen Isolate der Patienten, und fünf internationale Patienten (zwei aus Frankreich, zwei aus den Niederlanden und einer aus Tunesien) wurden für die Phagentherapie nach Brüssel überwiesen.
Enter Synthetische Biologie
Mit Beginn des dritten Jahrtausends wurden zunehmend Ansätze der Synthetischen Biologie entwickelt, um die Spezifität von Phagen und das Auftreten bakterieller Phagenresistenzen zu verringern (z. B., strukturgeleitetes Design) (Pires et al., 2016; Dunne et al., 2019). So wurden beispielsweise Hefe-basierte Plattformen für den Wechsel von Phagenschwanzfaserproteinen entwickelt, um hybride Phagen mit einem besser vorhersagbaren und erweiterten Wirtsspektrum zu entwickeln (Ando et al., 2015; Yosef et al., 2017), und gentechnische Strategien (z. B., CRISPR-Cas-Editing-Tools) wurden entwickelt, um andere Aspekte wie negative Patienten-Phagen-Interaktionen (z. B. Anti-Phagen-Immunreaktionen) (Brown et al., 2017), die potenzielle Entstehung und Verbreitung bakterieller Phagenresistenzmechanismen und die Freisetzung schädlicher bakterieller Inhaltsstoffe wie Endotoxine (Hwang et al., 2018) anzugehen. Synthetische Phagengenome mussten neu gebootet werden, um Phagen-Nachkommen zu produzieren (Barbu et al., 2016; Pires et al., 2016), und zwar durch die Transformation von Escherichia coli oder Listeria monocytogenes L-Formen (Kilcher et al., 2018) oder durch zellfreie Transkriptions-Translations-Systeme (TXTL) (Rustad et al., 2018). Westliche Regulierungsrahmen begannen allmählich, präzisen und personalisierten Phagentherapieansätzen unter Verwendung natürlich vorkommender Phagen (Pirnay et al., 2018), gentechnisch hergestellter Phagen (Dedrick et al., 2019) und synthetischer Phagen Rechnung zu tragen.
Die Entwicklung von Geräten zur Ad-hoc- und Vor-Ort-Produktion therapeutischer Phagen, wie Phage-BEAM, verlief nicht reibungslos, erinnerte Iverian. Zunächst war ein auf künstlicher Intelligenz (KI) basierendes In-silico-Phagen-Matching und -Design erforderlich. Deep Learning (Martorell-Marugán et al., 2019), eine Untergruppe des maschinellen Lernens, wurde für die Suche nach Verbindungen zwischen bakteriellen Genomen und infizierenden Phagengenomen gewählt, da es sich leichter auf eine größere Anzahl von Proben skalieren ließ. So erforderten Deep-Learning-Methoden keine sogenannte Merkmalsextraktion, die eine Annotation von Phagen- und Bakteriengenomen auf Gen-/Proteinebene erfordern würde und die Vorhersagen auf bestimmte bekannte Beziehungen zwischen Bakterien- und Phagenmerkmalen beschränken würde, wie z. B. Phagenschwanzfaserstrukturen, die an bestimmte bakterielle Zellwandrezeptoren binden. Die Kehrseite der Medaille bestand darin, dass eine kontinuierliche Versorgung mit riesigen Datenmengen erforderlich war, um lytische Phagengenome mit bakteriellen Wirtsgenomen zu verknüpfen, und genau hier drückte der Schuh. Die Ganzgenomsequenzierung hatte langsam Einzug in die Praxis der klinischen Mikrobiologie gehalten (American Academy of Microbiology, 2016), aber Forschungsinstitute und Pharmaunternehmen waren nicht daran interessiert, ihre Daten an eine einzige zentrale Datenbank zu übermitteln, und es fanden sich keine Investoren, die bereit und in der Lage waren, die verfügbaren Daten zu erwerben und/oder ausreichende Mengen neuer Daten zu produzieren. Ein zweites Hindernis, das es zu überwinden galt, war das Fehlen einer schnellen, zuverlässigen und erschwinglichen Synthese von großen DNA-Molekülen. Die ersten DNA-Synthesetechniken basierten auf der organischen Chemie und produzierten relativ kleine DNA-Moleküle. Die De-novo-Synthese von Phagengenomen erforderte die Assemblierung mehrerer Genomfragmente (Barbu et al., 2016; Pires et al., 2016; Lemire et al., 2018) in der Hefe Saccharomyces cerevisiae, die Verwendung von künstlichen Hefechromosomen (Ando et al., 2015) oder die chemische Assemblierung (Gibson et al., 2009). Die Entwicklung einer neuen Technik zur DNA-Synthese, die auf DNA-synthetisierenden Enzymen in Zellen des Immunsystems basiert (Palluk et al., 2018), erleichterte die Phagengenomsynthese. Schließlich mussten einige Hürden überwunden werden, um generische zellfreie Phagenproduktionssysteme zu entwickeln, die in der Lage sind, Phagen in hohen Titern zu produzieren und das gleiche Maß an bakterieller Infektiosität wie ihre natürlichen Analoga aufzuweisen.
Der Durchbruch
Das Hauptproblem bestand darin, dass es sich als sehr schwierig erwies, die riesigen Mengen an verknüpften Phagen- und Bakteriengenomsequenzen zu sammeln, die für die Deep-Learning-KI-Algorithmen erforderlich sind, um Phagensequenzen mit einem therapeutisch akzeptablen Maß an Genauigkeit vorherzusagen und/oder zu entwerfen. Iverian erinnerte sich, dass der eigentliche Durchbruch erfolgte, als die gemeinnützige Organisation “Phage XChange” ihre gleichnamige globale Phagen-Governance-Plattform ins Leben rief, um eine effiziente, standardisierte, nachhaltige und ethische Phagen-Lieferkette zu schaffen (Abbildung 2). Phage XChange bestand hauptsächlich aus einem KI-Modul und einem Distributed Ledger (DL) (Thiebes et al., 2020). Das KI-Modul der Plattform analysierte verknüpfte Phagen- und Bakteriengenome, um wirksame Phagen für Kunden vorherzusagen und zu entwickeln. Auf der Grundlage des Internets der Dinge (IoT) und von Big Data sowie von Informationen, die von internationalen Organisationen des öffentlichen Gesundheitswesens wie der Weltgesundheitsorganisation (WHO) und den nationalen Zentren für Seuchenkontrolle bereitgestellt wurden, sagte es außerdem voraus, welche bakteriellen Krankheitserreger am dringendsten behandelt werden müssen. Diese Daten lenkten das System auf die Isolierung und Charakterisierung der am dringendsten benötigten Phagen.
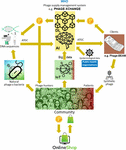
Abbildung 2. Vision, wie die Phagenlieferkette im Jahr 2035 organisiert sein könnte. AI, künstliche Intelligenz; ATGC, DNA-Sequenz; BEAM, bedside energized anti-microbial; DL, distributed ledger; IoT, Internet of Things; P, PhageCoin; WHO, World Health Organization.
Das DL-Modul der Plattform sorgte für einen ausreichenden, qualitativen und protokollierten Input von verknüpften Phagen-/Bakteriengenomsequenzen an das KI-Modul und dito für die Bereitstellung von Phagensequenzen an Kunden, in Übereinstimmung mit den Bestimmungen des Nagoya-Protokolls (Expert round table on acceptance and re-implementation of bacteriophage therapy et al, 2018). Die DL zeichnete alle Beteiligten (z. B. Datenlieferanten, Sequenzierungsdienste und Kunden), Transaktionen und Vertragsdetails unveränderbar auf. Außerdem wurden die genaue Qualität, die Spezifikationen und das Gewicht des gelieferten Materials erfasst. Ein Algorithmus stellte die Nicht-Redundanz fest und schätzte das Gewicht (z. B. die Virulenz und den Wirtsbereich der Phagen) und die Erwünschtheit des eingereichten Materials. Phagen, die auf neue bakterielle Krankheitserreger abzielen, waren natürlich am begehrtesten. Die meisten Patentfragen waren damit vom Tisch. Die DL fungierte als Zahlungsverwaltung, um sicherzustellen, dass alle Parteien rechtzeitig und fair bezahlt wurden. Eine Reihe von PhageCoins (die Kryptowährung der Plattform) wurde den Lieferanten in Abhängigkeit von der Qualität, dem Gewicht und der Erwünschtheit des gelieferten Materials zugewiesen. Kunden, die Vorhersageergebnisse (Phagensequenzen) über die DL extrahierten, zahlten einen Betrag an PhageCoins, der dem geschätzten Wert der Phagen entsprach. Diese PhageCoins wurden verwendet, um die DL aufrechtzuerhalten, einen ausreichenden und kontinuierlichen Zufluss von Material zu gewährleisten und die Daten über die Virulenz und den Wirtsbereich von Phagen zu erweitern (Zuordnung von Phagen zu Bakterien). Eine zusätzliche Finanzspritze und ein Anreiz, Material zu liefern, wurde bei Herstellern und Lieferanten von Waren aller Art gefunden. Mit den sofort verdienten PhageCoins konnten Phagenlieferanten online alle möglichen Produkte zu stark reduzierten Preisen kaufen, von Labor- und Schulausrüstung bis hin zu Sportartikeln. Diese Waren wurden durch Firmensponsoring zur Verfügung gestellt. Mehrere etablierte Unternehmen sponserten PhageXchange im Austausch gegen Steuererleichterungen, Werbung und das Image einer sozial verantwortlichen Marke. Das Gewicht des gelieferten Materials und damit auch sein Wert wurden anfangs unterbewertet, aber in regelmäßigen Abständen neu bewertet (Iteration), und den Lieferanten wurden mehr PhageCoins zugewiesen, wenn dies gerechtfertigt war. Obwohl die Plattform von Anfang an nützlich war, wurde sie erst dann wirklich erfolgreich, als sie unter den Schutz der WHO gestellt wurde, in Analogie zum weltweiten System der Rückverfolgbarkeit, Transparenz, Wachsamkeit und Überwachung von Arzneimitteln menschlichen Ursprungs (Warwick et al., 2013). Eine formelle Vereinbarung zwischen Phage XChange und der WHO stärkte das internationale Vertrauen in die langfristige Nachhaltigkeit der Plattform und den Schutz vor unethischer kommerzieller Ausbeutung. Die Suche nach wirksamen therapeutischen Phagen wurde bald zu einer Gemeinschaftsanstrengung, die darauf abzielte, die Krise der Antibiotikaresistenz zu lösen. Unabhängige “Phagenjäger”, Schulen, Pfadfindergruppen, Dörfer an den Ufern des Amazonas usw. isolierten Phagen und reichten sie bei Phage XChange ein, im Austausch gegen PhageCoins. Am Rande dieses Prozesses entwickelten verschiedene Unternehmen und Institutionen Peripheriegeräte und Dienstleistungen, wie z. B. Phagenisolierungskits und Sequenzierungs- und Phagensyntheseplattformen (z. B. das Phage-BEAM-Gerät). Im Vorgriff auf diese Geräte wurden Zwischenlösungen angeboten, bei denen die Phagen selbst über die DL gewonnen wurden.
Epilog
Dieser Ausblick auf die Zukunft der Phagentherapie bietet einen optimistischen Ausweg aus der Antibiotikaresistenzkrise. Die Ad-hoc- und Vor-Ort-Produktion synthetischer Phagen, verbunden mit einem globalen, gemeinschaftsbasierten Phagen-Management-System, erwies sich als willkommene und (für die Sozialversicherungssysteme) erschwingliche zusätzliche Waffe im Kampf gegen antibiotikaresistente bakterielle Infektionen. Es handelte sich jedoch nicht um eine Wunderwaffe, sondern um eine synergistische Ergänzung zu den etablierten antimikrobiellen Mitteln. Die sofortige und zellfreie Produktion von synthetischen Phagen, ob sie nun entwickelt wurden oder nicht, hatte erhebliche Vorteile gegenüber klassisch (in bakteriellen Wirten) produzierten natürlichen Phagen:
(i) Es bestand keine Notwendigkeit, physische therapeutische Phagenbanken zu unterhalten und die bakteriellen Isolate des Patienten und die passenden therapeutischen Phagen in die ganze Welt zu versenden.
(ii) Synthetische Phagen gegen Bakterien, die eminente Bedrohungen für die öffentliche Gesundheit darstellen, wie z. B. der Ausbruch von E. coli O104:H4 in Deutschland 2011 (Merabishvili et al., 2012), oder Bakterien, die (mutmaßlich) für Bioterrorismus verwendet werden (Joñczyk-Matysiak et al.,
(iii) Phagen gegen Bakterien, die potenziell tödliche Krankheiten verursachen, für die keine nicht-tödlichen Produktionswirtsstämme zur Verfügung stehen und deren Vermehrung bisher Biosicherheitsstufe-3-Vorkehrungen erforderte, könnten unter bsl-1-Bedingungen synthetisiert werden.
(iv) Wenn an Probenahmestellen keine Phagen isoliert werden konnten, beispielsweise weil die für die Isolierungstechniken verwendeten bakteriellen Wirtsstämme nicht für die gewünschten Phagen empfänglich waren, wurden (vorhergesagte) genomische Phagensequenzen, die aus metagenomischen Daten extrahiert wurden (Reyes et al, 2010; Amgarten et al., 2018), könnten zur Herstellung synthetischer Phagen verwendet werden.
(v) Synthetische Phagenpräparate enthielten keine (oder geringere Mengen an) Moleküle, die sich negativ auf Patienten auswirken könnten (z. B. Endotoxine).
(vi) Geräte wurden angepasst, um synthetische Phagen während einer ausgedehnten Weltraumreise und Weltraumkolonisation zu produzieren (Taylor und Sommer, 2005).
Es besteht eine geringe Chance, dass diese Vorhersagen eintreten. Es ist wahrscheinlich zu kurzsichtig zu glauben, dass eine gemeinschaftsbasierte Anstrengung, die von Organisationen des öffentlichen Gesundheitswesens unterstützt und von einer globalen, nachhaltigen und ethischen Plattform verwaltet wird, das Herzstück einer Lösung für die derzeitige weltweite Krise der Antibiotikaresistenz sein könnte. Einige Teile des vorgeschlagenen Systems, wie z. B. die zellfreie Herstellung synthetischer Phagen mit einem Gerät am Krankenbett, haben gute Chancen auf Verwirklichung, während andere Elemente, wie z. B. das Sponsoring durch Unternehmen, wahrscheinlich auf das Reich der Science-Fiction beschränkt bleiben werden. Sie können sagen, dass ich ein Träumer bin, also wecken Sie mich ruhig im Jahr 2035 auf, um mich mit der Realität zu konfrontieren!
Beiträge der Autoren
J-PP hat die Vision entwickelt und das Manuskript verfasst.
Finanzierung
Die Kosten für die Veröffentlichung wurden von der “Société Scientifique du Service Médical Militaire – Wetenschappelijke Vereniging van de Militaire Medische Dienst” übernommen.
Interessenkonflikt
Der Autor erklärt, dass die Forschung in Abwesenheit jeglicher kommerzieller oder finanzieller Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Der bearbeitende Herausgeber hat eine frühere Koautorenschaft mit dem Autor erklärt.
Danksagungen
Die persönliche Vision oder der Traum, der in diesem Manuskript entwickelt wurde, entstand als Ergebnis von Interaktionen mit vielen Forscherkollegen in den letzten 15 Jahren. Es ist unmöglich, sie alle zu nennen, aber es wäre auch nicht fair, alle Lorbeeren allein zu ernten. Daher habe ich mich entschlossen, einige von ihnen hier (in alphabetischer Reihenfolge) zu erwähnen, auch auf die Gefahr hin – oder besser: mit der Gewissheit -, dass ich einige wichtige Einflussnehmer vergessen habe: Joana Azeredo, Nata Bakuradze, Bob Blasdel, Dimitri Boeckaerts, Angus Buckling, Yves Briers, Pieter-Jan Ceyssens, Nina Chanishvili, Laurent Debarbieux, Sarah Djebara, Dorien Dams, Daniel De Vos, Quirin Emslander, Alan Fauconnier, Ville Friman, Andrzej Górski, Téa Glonti, Nino Grdzelishvili, Serge Jennes, Elene Kakabadze, Betty Kutter, Rob Lavigne, Cédric Lood, Alice Maestri, Khatuna Makalatia, Maya Merabishvili, Tobi Nagel, Thomas Rose, Patrick Soentjens, Michiel Stock, Rüdiger Trojok, An Van den Bossche, Mario Vaneechoutte, Gilbert Verbeken, und Kilian Vogele.
American Academy of Microbiology (2016). Applications of Clinical Microbial Next-Generation Sequencing: Report on an American Academy of Microbiology Colloquium Held in Washington, DC, in April 2015. Washington, DC: American Society for Microbiology.
Google Scholar
Amgarten, D., Braga, L. P. P., da Silva, A. M., and Setubal, J. C. (2018). MARVEL, ein Werkzeug zur Vorhersage von Bakteriophagen-Sequenzen in metagenomischen Bins. Front. Genet. 9:304. doi: 10.3389/fgene.2018.00304
PubMed Abstract | CrossRef Full Text | Google Scholar
Kilcher, S., Studer, P., Muessner, C., Klumpp, J., and Loessner, M. J. (2018). Gattungsübergreifendes Rebooten von massgeschneiderten, synthetischen Bakteriophagengenomen in L-form Bakterien. Proc. Natl. Acad. Sci. U.S.A. 115, 567-572. doi: 10.1073/pnas.1714658115
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemire, S., Yehl, K. M., and Lu, T. K. (2018). Phagenbasierte Anwendungen in der synthetischen Biologie. Annu. Rev. Virol. 5, 453-476. doi: 10.1146/annurev-virology-092917-043544
PubMed Abstract | CrossRef Full Text | Google Scholar
Luria, S. E., and Delbrück, M. (1943). Mutationen von Bakterien von Virusempfindlichkeit zu Virusresistenz. Genetics 28, 491-511.
Google Scholar
Martorell-Marugán, J., Tabik, S., Benhammou, Y., del Val, C., Zwir, I., Herrera, F., et al. (2019). “Deep Learning in omics data analysis and precision medicine,” in Computational Biology, ed. H. Husi (Brisbane: Codon Publications), 37-53. doi: 10.15586/computationalbiology.2019.ch3
PubMed Abstract | CrossRef Full Text | Google Scholar
Merabishvili, M., De Vos, D., Verbeken, G., Kropinski, A. M., Vandenheuvel, D., Lavigne, R., et al. (2012). Auswahl und Charakterisierung eines therapeutischen Bakteriophagen-Kandidaten, der den Escherichia coli-Stamm O104:H4 aus dem Ausbruch von 2011 in Deutschland lysiert. PLoS One 7:e52709. doi: 10.1371/journal.pone.0052709
PubMed Abstract | CrossRef Full Text | Google Scholar
Miêdzybrodzki, R., Borysowski, J., Weber-Da̧browska, B., Fortuna, W., Letkiewicz, S., Szufnarowski, K., et al. (2012). Klinische Aspekte der Phagentherapie. Adv. Virus Res. 83, 73-121. doi: 10.1016/B978-0-12-394438-2.00003-7
PubMed Abstract | CrossRef Full Text | Google Scholar
Palluk, S., Arlow, D. H., de Rond, T., Barthel, S., Kang, J. S., Bector, R., et al. (2018). De-novo-DNA-Synthese mit Polymerase-Nukleotid-Konjugaten. Nat. Biotechnol. 36, 645-650. doi: 10.1038/nbt.4173
PubMed Abstract | CrossRef Full Text | Google Scholar
Pires, D. P., Cleto, S., Sillankorva, S., Azeredo, J., and Lu, T. K. (2016). Gentechnisch veränderte Phagen: ein Überblick über die Fortschritte der letzten zehn Jahre. Microbiol. Mol. Biol. Rev. 80, 523-543. doi: 10.1128/MMBR.00069-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., De Vos, D., Verbeken, G., Merabishvili, M., Chanishvili, N., Vaneechoutte, M., et al. (2011). Das Paradigma der Phagentherapie: prêt-à-porter oder sur-mesure? Pharm. Res. 28, 934-937. doi: 10.1007/s11095-010-0313-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., Verbeken, G., Ceyssens, P. J., Huys, I., De Vos, D., Ameloot, C., et al. (2018). The magistral phage. Viruses 10:E64. doi: 10.3390/v10020064
PubMed Abstract | CrossRef Full Text | Google Scholar
Reyes, A., Haynes, M., Hanson, N., Angly, F. E., Heath, A. C., Rohwer, F., et al. (2010). Viren in der fäkalen Mikrobiota monozygoter Zwillinge und ihrer Mütter. Nature 466, 334-338. doi: 10.1038/nature09199
PubMed Abstract | CrossRef Full Text | Google Scholar
Rustad, M., Eastlund, A., Jardine, P., and Noireaux, V. (2018). Zellfreie TXTL-Synthese von infektiösem Bakteriophagen T4 in einer einzigen Reagenzglasreaktion. Synth. Biol. 3:ysy002.
Google Scholar
Schooley, R. T., Biswas, B., Gill, J. J., Hernandez-Morales, A., Lancaster, J., Lessor, L., et al. (2017). Entwicklung und Einsatz von personalisierten Bakteriophagen-basierten therapeutischen Cocktails zur Behandlung eines Patienten mit einer disseminierten resistenten Acinetobacter baumannii-Infektion. Antimicrob. Agents Chemother. 61:e0954-17. doi: 10.1128/AAC.02221-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Sulakvelidze, A., Alavidze, Z., and Morris, J. G. Jr. (2001). Bacteriophage therapy. Antimicrob. Agents Chemother. 45, 649-659.
Google Scholar
Suttle, C. A. (2013). Viren: Erschließung der größten Artenvielfalt der Erde. Genome 56, 542-544. doi: 10.1139/gen-2013-0152
PubMed Abstract | CrossRef Full Text | Google Scholar
Taylor, P. W., and Sommer, A. P. (2005). Auf dem Weg zu einer rationalen Behandlung von bakteriellen Infektionen während ausgedehnter Weltraumreisen. Int. J. Antimicrob. Agents 26, 183-187. doi: 10.1016/j.ijantimicag.2005.06.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiebes, S., Schlesner, M., Brors, B., und Sunyaev, A. (2020). Distributed ledger technology in genomics: a call for Europe. Eur. J. Hum. Genet. 28, 139-140. doi: 10.1038/s41431-019-0512-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiel, K. (2004). Altes Dogma, neue Tricks – Phagentherapie im 21. Jahrhundert. Nat. Biotechnol. 2, 31-36. doi: 10.1038/nbt0104-31
PubMed Abstract | CrossRef Full Text | Google Scholar
Vandersteegen, K., Mattheus, W., Ceyssens, P. J., Bilocq, F., De Vos, D., Pirnay, J.-P., et al. (2011). Mikrobiologische und molekulare Bewertung von Bakteriophagen ISP für die Bekämpfung von Staphylococcus aureus. PLoS One 6:e24418. doi: 10.1371/journal.pone.0024418
PubMed Abstract | CrossRef Full Text | Google Scholar
Verbeken, G., Pirnay, J. P., De Vos, D., Jennes, S., Zizi, M., Lavigne, R., et al. (2012). Optimierung des europäischen Rechtsrahmens für eine nachhaltige Bakteriophagentherapie in der Humanmedizin. Arch. Immunol. Ther. Exp. 60, 161-172. doi: 10.1007/s00005-012-0175-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Warwick, R. M., Chapman, J., Pruett, T. L., and Wang, H. (2013). Global einheitliche Kodierungssysteme für Medizinprodukte menschlichen Ursprungs. Bull. World Health Organ. 91, 314-314A. doi: 10.2471/BLT.12.116988
PubMed Abstract | CrossRef Full Text | Google Scholar
Yosef, I., Goren, M. G., Globus, R., Molshanski-Mor, S., and Qimron, U. (2017). Erweiterung des Wirtsbereichs von Bakteriophagenpartikeln für die DNA-Transduktion. Mol. Cell. 66, 721.e3-728.e3. doi: 10.1016/j.molcel.2017.04.025
PubMed Abstract | CrossRef Full Text | Google Scholar