ChIP-seq on hieno tekniikka, jonka avulla voimme tutkia proteiinin ja DNA:n välisiä fysikaalisia sitoutumisvuorovaikutuksia seuraavan sukupolven sekvensoinnin avulla. Tässä artikkelissa annan lyhyen katsauksen ChIP:hen ja esittelen kromatiinin immunoprecipitointisekvensointitekniikan (ChIP-seq), jossa yhdistetään ChIP ja seuraavan sukupolven sekvensointi.
Mitä on kromatiinin immunoprecipitointi?
Kromatiinin immunoprecipitointi (ChIP) mahdollistaa proteiinien sitoutumiskohtien määrittämisen DNA:lla. Kromatiini on DNA:n kompleksi, joka on pakattu histoniproteiinien kanssa nukleosomeiksi. ChIP:ssä hyödynnetään DNA:n ja siihen liittyvien proteiinien välille solujen tai kudoksen formaldehydifiksaatiossa syntyneitä palautuvia ristisidoksia. Fiksattu kromatiini leikataan fysikaalisesti, ja tiettyyn proteiiniin assosioituneet DNA-fragmentit saadaan selektiivisesti immunoprecipitoitua ja analysoitua. Analyysi voidaan tehdä lokuskohtaisesti PCR:n avulla, mutta yleisemmin ChIP:tä tutkitaan mikrosirujen (ChIP-chip) tai seuraavan sukupolven sekvensoinnin (ChIP-seq) avulla.
Miten ChIP-chip toimii?
ChIP-on-chip eli ChIP-chip yhdistää kromatiinin immunopresipitaation ja mikrosirujen analyysin. Vuonna 2001 Jason Lieb Pat Brownin laboratoriossa Stanfordissa julkaisi ensimmäisen ChIP-chip-artikkelin (Lieb 2001). Tässä menetelmässä DNA-fragmentit, jotka saostuvat tietyn proteiinin kanssa, levitetään mikrosirulle analysoitavaksi. Näin saadaan kokonaiskuva siitä, mihin proteiini sitoutuu, toisin kuin vain yksittäisten sitoutumiskohtien tutkimisessa PCR:llä. Vaikka tämä oli vallankumouksellinen lähestymistapa, tekniikkaa rajoittaa käytetty array-tekniikka. Ensinnäkin tarvitaan tutkittavaa genomia vastaava mikrosiru, ja 2000-luvun alussa ja puolivälissä näin ei useinkaan ollut. Toiseksi näitä mikrosiruja rajoittaa referenssigenomin laatu ja kyky suunnitella koettimia, jotka toimivat mikrosirulla. Kolmanneksi matriisit ovat rajoitettuja sen suhteen, kuinka suuri osa genomista voidaan edustaa, ja tavallisesti ne kattavat genomin 100 tai 1000 bp:n resoluutiolla. Ongelmia aiheuttavat myös ChIP-DNA-fragmenttien amplifikaatioharha, array-datan normalisointi ja array-alustojen vertailukelpoisuus.
Miten ChIP-seq toimii?
Kromatiini-immunoprecipitointisekvensoinnissa eli ChIP-seq:ssä yhdistetään ChIP:n ja seuraavan sukupolven sekvensoinnin yhdistelmä (Barski 2007, Johnson 2007). ChIP-seq-protokollat on mukautettu ChIP-chip-menetelmistä: proteiinit ristisidotaan niihin sitoutuneeseen DNA:han formaldehydikäsittelyllä, solut homogenisoidaan ja kromatiini leikataan ja immunoprecipitoidaan vasta-aineisiin sitoutuneilla magneettihelmillä. Immunoprecipitoitua DNA:ta käytetään sitten syötteenä seuraavan sukupolven sekvensointikirjaston esivalmisteluprotokollassa, jossa se sekvensoidaan ja analysoidaan DNA:n sitoutumiskohtien osalta. Alla olevassa kuvassa on yhteenveto ChIP-seq-työnkulusta ja esimerkki ChIP-seq-tuloksista (jäljennetty Dominic Schmidtin (Schmidt 2009) luvalla).
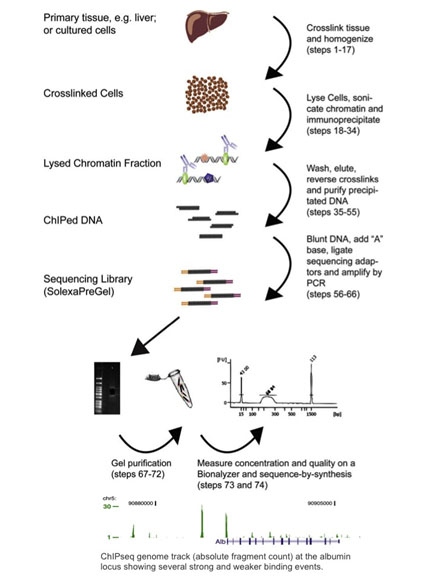
Vaikka suurin osa tähän mennessä julkaistuista noin 400:sta artikkelista on analysoitu Illumina-alustalla, ChIP-seq voidaan suorittaa millä tahansa seuraavan sukupolven sekvenssilaitteella (Wold 2008). ChIP-seq on otettu laajalti käyttöön sen jälkeen, kun siitä raportoitiin ensimmäisen kerran vuonna 2007. Itse asiassa se on syrjäyttänyt ChIP-Chipin lähes kokonaan, koska se mahdollistaa genominlaajuisen analyysin eikä sillä ole edellä käsiteltyjä rajoituksia.
Mitä ChIP-seq:lla voi oikeastaan tehdä
ChIP-seq on tehokas ja monipuolinen työkalu, ja kirjallisuudessa on monia hyviä esimerkkejä ChIP-seq:n käytöstä. Olen poiminut pari suosikkiani (johtamassani ydinlaitoksessa tehdyistä töistä) havainnollistamaan, mitä kaikkea on mahdollista, ja lisännyt esimerkkejä, joissa ChIP-seq on innoittanut uusien menetelmien kehittämiseen:
- Antoni Hurtado, et al. suorittivat FoxA1:n “pioneeritekijän” tyrmäyksen (knock-down), jonka tuloksena estrogeenireseptorin (ER) sitoutuminen väheni yli 50 %:lla tunnetuista ER:n sitomiskohdista. He osoittivat, että FoxA1 on tärkeä ER-välitteisen transkription säätelijä, mikä viittaa siihen, että se voi olla uusi ja tärkeä terapeuttinen kohde rintasyövässä (Hurtado 2011).
- Dominic Shmidt, ym. käyttivät ChIP-seq:ia transkriptiotekijöiden sitoutumisen evoluution tutkimiseen. He keskittyivät CEBPA:n ja HNF4:n sitoutumiseen viiden selkärankaisen lajin: ihmisen, hiiren, koiran, opossumin ja kanan maksakudoksessa. ChIP-chip-menetelmä olisi ollut lähes mahdoton, kun otetaan huomioon eri lajit ja koettimien suunnittelun monimutkaisuus (Schmidt 2010).
ChIP-seq-menetelmän jatkomuutokset ovat johtaneet useisiin uusiin menetelmiin, kuten RNA-proteiinien vuorovaikutusanalyyseihin tarkoitettuun ChIP-seq-menetelmään sekä DNase-seq- ja FAIRE-seq-menetelmiin, joita molempia käytetään DNA:n säätelyalueiden tunnistamiseen.
Yhteenvetona voidaan todeta, että ChIP-seq on kehittynyt tekniikka, joka on parempi kuin ChIP-chip, ja sitä voivat käyttää lähes kaikki ryhmät, jotka ovat kiinnostuneita DNA:proteiinien vuorovaikutusanalyysistä. Miten voisit käyttää ChIP-seq:tä tutkimuksessasi?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova & Marra, Applications of next-generation sequencing technologies in functional genomics. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Onko tästä ollut apua? Jaa sitten verkostollesi.