- Introducción
- Materiales y Métodos
- Muestras de Estudio
- Extracciones de ADN y ARN genómico
- Preparación de la biblioteca y secuenciación
- Captura de ADNg utilizando sondas IDT Xgen® Lockdown® y secuenciación de una sola molécula
- Captura de ADNc utilizando sondas IDT Xgen® Lockdown® y secuenciación de isoformas de una sola molécula (Iso-Seq)
- Análisis de ADNg
- Análisis de Variantes Cortas y Fases
- Clasificación y determinación de haplotipos para la región rica en CT
- Análisis de isoformas
- Calificación de SNP de isoformas
- Resultados
- La captura de ADNg dirigido identificó variaciones conocidas y novedosas
- La captura de ADNc dirigida identificó nuevos sitios de inicio y final
- El ADNc de longitud completa permite la información de fase a nivel de isoforma
- Discusión
- Disponibilidad de datos
- Contribuciones de los autores
- Financiación
- Declaración de conflicto de intereses
- Agradecimientos
- Material complementario
Introducción
Los programas transcripcionales y postranscripcionales controlan los niveles de expresión de los genes y/o la producción de múltiples isoformas distintas de ARNm, y los cambios en estos mecanismos dan lugar a una desregulación de la expresión de los genes y a perfiles de expresión diferenciales. La regulación transcripcional y postranscripcional aberrante de los genes es abundante en los tejidos del sistema nervioso humano y contribuye a las diferencias fenotípicas dentro de los individuos y entre ellos, tanto en la salud como en la enfermedad.
La desregulación de la expresión de la alfa-sinucleína se ha implicado en la patogénesis de las sinucleinopatías, en particular la enfermedad de Parkinson (EP) y la demencia con cuerpos de Lewy (DCL). Mientras que el papel de la sobreexpresión de SNCA en las sinucleinopatías, principalmente en la EP, ha sido bien establecido, aquí nos centramos en la determinación del repertorio completo de isoformas de transcripción de SNCA en diferentes sinucleinopatías. Anteriormente, se han descrito varias isoformas de transcripción SNCA diferentes para el gen SNCA, surgidas del splicing alternativo, los sitios de inicio de la transcripción (TSS) y la selección de los sitios de poliadenilación (McLean et al., 2012; Xu et al., 2014). El splicing alternativo de los exones codificantes da lugar a SNCA 140, SNCA 112, SNCA 126 y SNCA 98, dando lugar a cuatro isoformas proteicas (Beyer y Ariza, 2012). Los TSSs alternativos del gen SNCA dan lugar a cuatro 5′UTRs diferentes, y la selección alternativa de diferentes sitios de poliadenilación determina tres longitudes principales del 3′UTR, sin impacto en la composición del producto proteico (Beyer y Ariza, 2012). Nuestro objetivo general es obtener nuevos conocimientos sobre la contribución de las diferentes especies de ARNm de SNCA, conocidas y nuevas, a la patogénesis y la heterogeneidad de las sinucleinopatías.
Hasta la fecha, la mayoría de los estudios han utilizado tecnologías de secuenciación de lectura corta para interrogar a la complejidad del transcriptoma en los cerebros humanos. La disponibilidad de tecnologías de lectura larga de tercera generación proporciona una imagen sin precedentes y casi completa de las estructuras de las isoformas. Sin embargo, la secuenciación de transcripción de lectura larga existente para los genes de enfermedades humanas ha utilizado un enfoque basado en amplicones (Treutlein et al., 2014; Kohli, 2017; Tseng et al., 2017). Si bien este enfoque ha tenido éxito en la identificación de empalmes alternativos complejos en genes de enfermedades humanas, está limitado al diseño del cebador de PCR y no descubrirá los sitios de inicio y final alternativos. El enriquecimiento selectivo, por ejemplo mediante el uso de sondas IDT, puede proporcionar una visión completa de las isoformas de los genes de interés a un bajo coste de secuenciación. Además, las lecturas de transcripción de longitud completa de alta precisión permiten el haplotipado específico de isoformas.
Aquí, presentamos el primer estudio conocido que utiliza la captura dirigida de ADNg y ADNc de la región del gen SNCA utilizando la secuenciación PacBio SMRT. La región del gen SNCA tiene una longitud de ~114 kb y consta de seis exones con longitudes de transcripción de alrededor de 3 kb. Multiplexamos 12 muestras de cerebro humano de EP, DCL y muestras de control normal y secuenciamos la biblioteca de ADNg y ADNc en el sistema PacBio Sequel. Describimos los análisis bioinformáticos utilizados para identificar SNPs, indels, y repeticiones cortas en tándem para la captura de gDNA, y el haplotyping a nivel de isoforma para los datos de cDNA. Demostramos que la captura dirigida es una forma rentable de estudiar conjuntamente la variación genómica y el splicing alternativo en un gen neuronal relacionado con la enfermedad.
Materiales y Métodos
Muestras de Estudio
La cohorte de estudio (N = 12) consistió en individuos con tres diagnósticos neuropatológicos confirmados por autopsia: (1) EP (N = 4); (2) DCL (N = 4); y (3) sujetos clínica y neuropatológicamente normales (N = 4). Los tejidos cerebrales de la corteza frontal fueron obtenidos a través del Banco de Cerebros Kathleen Price Bryan (KPBBB) en la Universidad de Duke, el Programa de Donación de Cerebros y Cuerpos del Instituto de Investigación Banner Sun Health (Beach et al., 2015), y el Centro de Envejecimiento y Enfermedad de Alzheimer Layton en la Universidad de Salud y Ciencias de Oregón. Los fenotipos neuropatológicos se determinaron en el examen postmortem siguiendo métodos estándar bien establecidos siguiendo el método y las recomendaciones de práctica clínica de McKeith y colegas (McKeith et al., 1999, 2005). La densidad de la patología LB (en un conjunto estándar de regiones cerebrales) recibió puntuaciones de leve, moderada, grave y muy grave. Las muestras de estudio dentro de cada grupo de diagnóstico, EP y DCL, fueron cuidadosamente seleccionadas de manera que la gravedad de los fenotipos clinicopatológicos fuera similar dentro de cada patología. Todos los cerebros mostraron cuerpos de Lewy (LB) en el tronco cerebral, límbicos y neocorticales, mientras que la EP mostró puntuaciones de McKeith de severas a muy severas en la subnigra y la amígdala. Todos los cerebros indican que no hay EA según los criterios del CERAD y el estadio de Braak y Braak = II. Las muestras de cerebros neurológicamente sanos se obtuvieron a partir de tejidos postmortem de sujetos clínicamente normales que fueron examinados, en la mayoría de los casos, en el plazo de un año después de la muerte y que no presentaban ningún trastorno cognitivo ni parkinsonismo, así como hallazgos neuropatológicos insuficientes para diagnosticar la EP, la enfermedad de Alzheimer (EA) u otros trastornos neurodegenerativos. Todas las muestras eran de raza blanca. Los datos demográficos y la neuropatología de estos sujetos se resumen en la Tabla Suplementaria 1. El proyecto fue aprobado por la Junta de Revisión Institucional (IRB) de Duke, que proporcionó una aprobación ética. Los métodos se llevaron a cabo de acuerdo con las directrices y reglamentos pertinentes.
Extracciones de ADN y ARN genómico
El ADN genómico se extrajo de los tejidos cerebrales mediante el protocolo estándar de Qiagen (Qiagen, Valencia, CA). El ARN total se extrajo de las muestras de cerebro (100 mg) utilizando el reactivo TRIzol (Invitrogen, Carlsbad, CA) seguido de la purificación con un kit RNeasy (Qiagen, Valencia, CA), siguiendo el protocolo del fabricante. La concentración de ADNg y ARN se determinó espectrofotométricamente, y la calidad de las muestras de ARN y la ausencia de degradación significativa se confirmaron mediante mediciones del número de integridad del ARN (RIN, Tabla suplementaria 1) utilizando un bioanalizador Agilent.
Preparación de la biblioteca y secuenciación
Captura de ADNg utilizando sondas IDT Xgen® Lockdown® y secuenciación de una sola molécula
Aproximadamente 2 μg de cada muestra de ADNg se cortaron a 6 kb utilizando el Covaris g-TUBE y se ligaron con adaptadores con código de barras. Se introdujo un conjunto equimolar de la biblioteca de ADNg con código de barras de 12 complejos (2 μg en total) en la captura basada en sonda con un panel de genes SNCA diseñado a medida.
Se construyó una biblioteca SMRTBell utilizando 626 ng de ADNg capturado y reamplificado1.
Captura de ADNc utilizando sondas IDT Xgen® Lockdown® y secuenciación de isoformas de una sola molécula (Iso-Seq)
Aproximadamente 100-150 ng de ARN total por reacción se transcribieron de forma inversa utilizando el kit de síntesis de ADNc Clontech SMARTer y 12 oligo dT con código de barras específico para la muestra (con secuencias de código de barras PacBio 16mer, ver Métodos Suplementarios). Se procesaron tres reacciones de transcripción inversa (RT) en paralelo para cada muestra. Se utilizó la optimización de la PCR para determinar el número óptimo de ciclos de amplificación para las reacciones de PCR a gran escala posteriores. Se utilizó un único cebador (cebador IIA del kit Clontech SMARTer 5′ AAG CAG TGG TAT CAA CGC AGA GTA C 3′) para todas las reacciones de PCR posteriores a la RT. Los productos de la PCR a gran escala se purificaron por separado con perlas 1X AMPure PB, y se utilizó el bioanalizador para el control de calidad. Se introdujo en la captura basada en sondas un pool equimolar de la biblioteca de ADNc con código de barras de 12 complejos (1 μg en total) con un panel de genes SNCA diseñado a medida.
Se construyó una biblioteca SMRTBell utilizando 874 ng de ADNc capturado y reamplificado2. Se secuenció una célula SMRT 1M (película de 6 h) en la plataforma PacBio Sequel utilizando química 2.0.
Análisis de ADNg
La secuenciación de los datos de ADNg con código de barras se realizó en tres células SMRT 1M utilizando química 2.0. Los datos se demultiplexaron ejecutando la aplicación Demultiplex Barcodes en PacBio SMRT Link v6.0.
Análisis de Variantes Cortas y Fases
Se generaron lecturas de Secuencia de Consenso Circular (CCS) utilizando SMRT Analysis 6.0 a partir de cada conjunto de datos demultiplexados y se alinearon con el genoma de referencia hg19 utilizando minimap2. Los duplicados de PCR de la amplificación posterior a la captura se identificaron mediante el mapeo de puntos finales y se etiquetaron utilizando un script personalizado. Las variantes cortas se llamaron utilizando GATK4 HaplotypeCaller (GATK4 HC) (Poplin et al., 2018). Después de una primera pasada de filtrado utilizando la profundidad de cobertura y las métricas de calidad, las variantes se inspeccionaron manualmente en IGV3. Si las variantes no entraban en fase con los SNP cercanos, se filtraron manualmente. Los sitios de variantes que pasaron la curación manual se utilizaron junto con las alineaciones CCS deduplicadas para el desfase respaldado por lectura con WhatsHap (Martin et al., 2016).
Clasificación y determinación de haplotipos para la región rica en CT
Se extrajeron las subsecuencias alineadas con chr4: 90742331-90742559 (hg19) para cada muestra. Tras inspeccionar la distribución del tamaño de estas subsecuencias, se agruparon por tamaño y similitud de secuencia utilizando una combinación de python y MUSCLE (Edgar, 2004), y se generó una secuencia de consenso de forma independiente para cada grupo.
Las secuencias de comandos personalizadas y los flujos de trabajo se describen con más detalle en https://github.com/williamrowell/Long-reads-Sequencing-of-SNCA-in-Diseases.
Análisis de isoformas
La secuenciación de los datos de ADNc con código de barras se realizó en un SMRT Cell 1M en el sistema PacBio Sequel utilizando química 2.0. El análisis bioinformático se llevó a cabo utilizando la aplicación IsoSeq3 en el PacBio SMRT Analysis v6.0.0 para obtener secuencias de isoformas de alta calidad y de longitud completa (ver Métodos Suplementarios para más información).
Calificación de SNP de isoformas
Las lecturas de longitud completa asociadas con las 41 isoformas finales de las 12 muestras se alinearon con el genoma hg19 para crear un pileup. Se excluyeron las bases con QV inferior a 13. A continuación, en cada posición con una cobertura de al menos 40 bases, se aplicó una prueba exacta de Fisher con corrección de Bonferroni con un corte de p de 0,01. Sólo se llamaron los SNPs de sustitución que no estaban cerca de regiones homopolímeras (tramos de 4 o más del mismo nucleótido). Después de la llamada de SNP, el genotipo de cada muestra se determinó contando el número de lecturas de longitud completa (FL) específicas de la muestra. Si una muestra tenía 5+ lecturas FL que apoyaban tanto la base de referencia como la alternativa, era heterocigota. Si una muestra tenía 5+ lecturas FL que apoyaban un alelo y menos de 5 lecturas FL para el otro, era homocigota. En caso contrario, no era concluyente. Los guiones están disponibles en: https://github.com/Magdoll/cDNA_Cupcake.
Resultados
Diseñamos sondas personalizadas para el gen SNCA y realizamos la captura dirigida tanto del gDNA como del cDNA en una biblioteca multiplexada que consistía en 12 muestras de cerebro humano de EP, DCL y controles normales (Figura 1, Tabla Suplementaria 1). Las bibliotecas de gDNA y cDNA fueron secuenciadas en la plataforma PacBio Sequel. El análisis bioinformático se realizó utilizando el software PacBio seguido de un análisis personalizado.
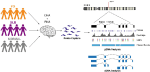
Figura 1. Presentación esquemática del diseño del estudio. Se extrajeron materiales de ADN y ARN de tejidos cerebrales postmortem de pacientes de la enfermedad de Parkinson, de la demencia con cuerpos de Lewy y de grupos de control. Se hicieron bibliotecas de ADNg y ADNc mediante hibridación con sonda y se secuenciaron en el sistema PacBio Sequel. El análisis se llevó a cabo utilizando el software PacBio y otras herramientas existentes.
La captura de ADNg dirigido identificó variaciones conocidas y novedosas
Después de generar secuencias circulares de consenso (CCS) y eliminar los duplicados de la PCR (Métodos suplementarios), obtuvimos una cobertura media única de 16 a 71 veces de la región del gen SNCA. Las lecturas CCS tenían una longitud media de inserción de 2,9 kb y una precisión media de lectura del 98,9%. Con la excepción de una región de 5 kb descubierta intencionadamente por las sondas debido a la presencia de elementos LINE (hg19 chr4: 90697216-90702113) y una región de 2,1 kb de alto contenido de GC alrededor del exón 1, hubo suficiente cobertura para genotipar ambos haplotipos para cada una de las 212 muestras (Figura 2, Figura suplementaria 1).

Figura 2. Captura de ADNg dirigido y desfase. Un ejemplo que muestra una muestra de cada condición. La pista superior muestra una de las isoformas SNCA, seguida de la cobertura de ADNg para las tres muestras. La pista de variantes muestra cada SNP y está codificada por colores para heterocigotos (púrpura), homocigotos alternativos (naranja) y homocigotos de referencia (gris). Los bloques de fase se muestran en azul claro. La pista inferior muestra las ubicaciones de las sondas de captura. La región de abandono en el diseño de la sonda se debe a dos elementos LINE en el centro del intrón 4. Para la cobertura de ADNg y la información de fase de las 12 muestras, véase las Figuras Suplementarias.
Usando GATK4 HC, el filtrado basado en la calidad y la curación manual, identificamos 282 SNPs y 35 indels, incluyendo 8 SNPS y 13 indels que no se encuentran en dbSNP (dbSNP Build ID: human_9606_b150_GRCh37p13) (Tabla Suplementaria 2). No se identificaron variantes en la región codificante de SNCA, aunque se identificaron ocho variantes en regiones no traducidas. La mayoría de las variantes identificadas, incluyendo varias repeticiones cortas en tándem (STR), caen dentro de los intrones 2, 3 y 4.
Hemos descrito previamente una región rica en CT altamente polimórfica en el intrón 4 de SNCA con cuatro haplotipos observados (Lutz et al., 2015). Aunque esta región altamente repetitiva y estructuralmente variable resultó difícil de genotipar con GATK4 HC, pudimos construir secuencias de consenso para las 12 muestras y observamos los 4 haplotipos descubiertos anteriormente (Figura suplementaria 2). Además, identificamos un nuevo STR en el intrón 4 consistente en una unidad de tres bases repetida 16 veces en la referencia. Dentro de las 12 muestras, identificamos tres haplotipos, con 9, 12 y 15 copias de la unidad de repetición TTG. GATK HC genotipó correctamente todos ellos, excepto un haplotipo para PD-4, que tenía una cobertura bastante baja en esta región. Sin embargo, con los datos dados para esta muestra, el genotipo puede determinarse por inspección visual (Tabla 1).

Tabla 1. Una nueva repetición de triplete en tándem en el intrón 4 (chr4: 90713442).
Utilizamos las variantes cortas detectadas por GATK HC junto con la herramienta de desfase basada en lecturas WhatsHap (Martin et al., 2016) para desfasar las lecturas CCS a través del locus, con un rango de éxito impulsado principalmente por la densidad de variantes heterocigotas sobre el locus. Las muestras PD-1, PD-4, N-4, DLB-1 y DLB-4 tenían tramos largos de baja heterocigosidad, con muy pocos bloques de fase cortos, mientras que las otras muestras produjeron bloques de fase que iban de 7 a 18 veces la longitud media de la lectura, hasta 54 kb (Figura Suplementaria 3).
La captura de ADNc dirigida identificó nuevos sitios de inicio y final
Procesamos los datos de ADNc de PacBio (Iso-Seq) utilizando el software de análisis SMRT de PacBio. Tras mapear los datos de Iso-Seq a hg19 y eliminar los artefactos (Tabla Suplementaria 3, Figura Suplementaria 4), obtuvimos un conjunto final de 41 isoformas SNCA (Figura 3). Todas las isoformas finales tienen todos los sitios de empalme canónicos (GT-AG o GC-AG) y están respaldadas por un total de 20 o más lecturas de longitud completa. La mayoría de las isoformas (28 de 41) tienen los seis exones y sólo difieren en el uso de sitios de inicio 5′ alternativos y en las longitudes de 3′ UTR. Las longitudes de 3′ UTR variaron entre 300 y 2,6 kb. El uso de sitios de inicio 5′ alternativos muy diversos en SNCA es conocido; lo que es menos conocido es la longitud variable de 3′ UTR, que se había estudiado previamente utilizando datos de RNA-seq que no resolvían las estructuras de las isoformas de longitud completa (Rhinn et al., 2012). Los datos de Iso-Seq muestran que la longitud variable 3′ UTR parece emparejada con todas las combinaciones posibles de sitios de inicio 5′ sin acoplamiento preferencial. Casi ninguna de la variabilidad en el sitio de inicio y final cambia el marco de lectura abierto predicho (Figura Suplementaria 5) y se predice que se traduce en la secuencia canónica de 141 aminoácidos.

Figura 3. Las isoformas de SNCA capturadas mediante Iso-Seq dirigida identifican nuevos sitios de inicio y finalización. La mayor parte de la complejidad de las isoformas proviene del uso combinatorio de longitudes alternativas del 3′ UTR y del exón 1, con unos pocos sitios de empalme alternativos encontrados en el exón 1 (verde), 2 (rojo) y 4 (azul). Todas las uniones tienen sitios de empalme canónicos. Identificamos cinco isoformas que se saltan el exón 5 y dos isoformas que se saltan el exón 3. También identificamos nuevos sitios de inicio (naranja) y final (púrpura) en el intrón 4. Los SNPs llamados están marcados en púrpura.
Además, validamos las uniones nuevas (pero canónicas) utilizando datos de unión de lectura corta disponibles públicamente. La base de datos Intropolis (v1, https://github.com/nellore/intropolis) combina más de 21.000 RNA-seq disponibles públicamente. Debido al alto volumen de datos de unión apoyados por una sola lectura corta, para este estudio, requerimos un mínimo de 10 lecturas cortas de apoyo (combinadas de todos los >21.000 conjuntos de datos de RNA-seq) para confirmar nuestras nuevas uniones Iso-Seq. Con la excepción de las nuevas uniones para PB.1016.253 y PB.1016.296 (Figura 3), todas las demás uniones nuevas están respaldadas por el conjunto de datos de Intropolis. Curiosamente, estas nuevas uniones tienen un apoyo significativamente menor a las lecturas cortas que las uniones anotadas en el Gencode. Por ejemplo, las dos nuevas uniones en PB.1016.139 introducidas por el nuevo exón están respaldadas por 2.519 y 44 recuentos de lecturas cortas de Intropolis, respectivamente, mientras que las otras cuatro uniones conocidas están respaldadas por más de 1 millón de recuentos de lecturas cortas. Esto demuestra el poder del enriquecimiento dirigido utilizando la secuenciación del transcriptoma completo para detectar isoformas raras y nuevas.
Observamos dos isoformas con omisión del exón 3 (SNCA126) y cinco isoformas con omisión del exón 5 (SNCA112). Una vez más, la diversidad de empalme en estos dos grupos de omisión de exón proviene principalmente del uso diverso de sitios de inicio 5′ alternativos y la longitud variable de 3′ UTR. La predicción del ORF muestra que la omisión del exón 3 o del exón 5 acorta el ORF pero mantiene el marco de lectura. Tres isoformas tienen nuevos sitios de inicio 3′ localizados en el intrón 4. La predicción del ORF muestra que esto da lugar a un producto proteico truncado.
Identificamos un sitio de inicio 5′ previamente no anotado situado en el intrón 4 (hg19 chr4: 90692548-90693045, Figura 3). Las tres isoformas asociadas a este nuevo inicio consisten en el nuevo sitio de inicio, el exón 5 y longitudes variables de 3′ UTR. Curiosamente, mientras que los datos de lecturas cortas descargados públicamente de GTEx y Sandor et al. (2017) y los datos de picos de CAGE (FANTOM5) no apoyaron este sitio de inicio novedoso, un conjunto de datos públicos recientes de ARN directo de NA128784 contenía solo una transcripción SNCA que confirmaba este sitio de inicio alternativo. Además, la nueva unión entre el exón 5 y el nuevo sitio de inicio está confirmada por los datos de unión de lectura corta de Intropolis. Curiosamente, se predice que este nuevo sitio de inicio 5′ introduce nuevos péptidos mientras mantiene el marco de lectura en el exón 5.
También identificamos tres transcripciones SNCA con nuevos sitios finales (Figura 3). Dos isoformas (PB.1016.383, PB.1016.384) utilizaron una 3′ UTR extendida en el exón 4, mientras que la tercera isoforma (PB.1016.381) utilizó un nuevo exón 3′ en el intrón 4. Las nuevas uniones entre el último exón novedoso y el exón anterior están respaldadas por datos públicos de unión de lecturas cortas (Intropolis). Los nuevos 3′ UTRs resultan en una predicción de ORF truncado.
Usando el recuento normalizado de lecturas de longitud completa como un proxy para la abundancia de isoformas, encontramos que una de las isoformas canónicas de SNCA (PB.1016.131) es la más abundante, con una abundancia del 50-60% en todas las muestras de sujetos (Tabla Suplementaria 4). Además, agrupamos las 41 isoformas según sus patrones de empalme (Tabla 2). Las isoformas que tienen los seis exones representan el 95-97% de la abundancia. Estudios anteriores han mostrado un marcado aumento de la expresión de las isoformas a las que les falta el exón 3 (SNCA126) en la corteza frontal de las muestras de DCL en comparación con las normales (Beyer et al., 2008); nuestros recuentos agregados de isoformas muestran que tres de las muestras de DCL tienen un nivel de recuento ligeramente elevado en comparación con las muestras normales, así como las variantes de SNCA112 (omisión del exón 5) para la EP y la DCL frente a las muestras normales.

Tabla 2. Abundancia de isoformas SNCA para cada muestra, agregada por patrones de empalme.
El ADNc de longitud completa permite la información de fase a nivel de isoforma
Llamamos a los SNPs utilizando el ADNc apilando todas las lecturas de longitud completa de las 12 muestras para llamar a las variantes (véase la sección “Métodos”). Se llamaron un total de cuatro SNPs y todos estaban previamente anotados en dbSNP (Tabla 3, Figura 3). Los cuatro SNPs están localizados en regiones no-CDS, uno en el 3′ UTR (exón 6), uno en el intrón 4, y dos en el 5′ UTR (exón 1). El SNP de la 3′ UTR (chr4: 90646886) sólo está cubierto por isoformas con una 3′ UTR de al menos ~1 kb de longitud, por lo que no todas las isoformas canónicas cubren este SNP. El SNP del intrón 4 (chr4: 90743331) sólo está cubierto por las nuevas isoformas alternativas del extremo 3′ (PB.1016.383, PB.1016.384) y no está conectado a ninguno de los otros SNPs. Los dos SNPs del 5′ UTR (chr4: 90757312 y chr4: 90758389) están cubiertos por dos usos mutuamente excluyentes del exón 1 y, por lo tanto, tampoco están conectados.

Tabla 3. Información del SNP del cDNA.
Nuestro enfoque actual se limita a llamar sólo a las variantes de sustitución en las regiones transcritas con suficiente cobertura. La comparación de la lista de nuestros SNPs con la anotación hg19 dbSNP muestra que la mayoría de los SNPs o variantes omitidas tenían una frecuencia inferior al 1% en la población, no eran sustituciones de un solo nucleótido o eran adyacentes a regiones de baja complejidad. Por ejemplo, el rs77964369 (chr4: 90646532) tiene una frecuencia del 50/50 de T/A; sin embargo, esta T es adyacente a un tramo de 11 As genómicas aguas abajo. La inspección manual de la pila de lecturas Iso-Seq, que tiene ~1.300 lecturas en este sitio, no sugiere evidencia de variación al menos entre nuestras 12 muestras.
Usando las lecturas específicas de la muestra, llamamos al genotipo de cada muestra en cada localización del SNP (Tabla 3). Aparte de que PD-2 tiene muy pocas lecturas y no es concluyente para los cuatro SNPs, pudimos determinar el genotipo de la mayoría de las otras muestras. En particular, DLB-3 fue la única muestra que es heterocigota en todas las localizaciones del SNP. Por lo demás, no observamos ningún patrón específico de la condición de preferir un genotipo a otro.
Discusión
Describimos el primer estudio que utiliza el enriquecimiento dirigido del gen SNCA en bibliotecas de ADNg y ADNc multiplexadas para estudiar enfermedades neurológicas utilizando la secuenciación de lectura larga. Las largas longitudes de lectura del sistema PacBio Sequel facilitaron la secuenciación del repertorio de isoformas de transcripción de longitud completa del gen SNCA. Revelamos la diversidad en el uso de sitios alternativos de inicio 5′ y longitudes variables de 3′ UTR y observamos eventos conocidos de omisión de exón, como la deleción del exón 3 (SNCA126) y la deleción del exón 5 (SNCA112). Además, se identificaron nuevos sitios alternativos de inicio y finalización dentro del gran intrón 4 que se prevé que se traduzcan en nuevas proteínas. Es probable que la alta profundidad de la cobertura de secuenciación de la captura dirigida, en combinación con la capacidad de secuenciar transcritos completos, nos haya permitido detectar estas isoformas no descritas previamente.
El significado biológico y patológico de las diferentes isoformas de la proteína SNCA aún no se ha descubierto completamente. Sin embargo, se han asociado isoformas específicas de SNCA de modificación postraduccional y de splicing con propensiones a la agregación intracelular (Kalivendi et al., 2010) y se expresan de forma diferente en las sinucleinopatías humanas (Beyer et al., 2008; Beyer y Ariza, 2012). Los estudios sobre la modificación postraduccional de SNCA mostraron que los cuerpos de Lewy, el sello patológico de las sinucleinopatías, contienen abundante SNCA fosforilada, nitrada y monoubiquitinada (Kim et al., 2014). También se han estudiado los efectos de la modificación post-transcripcional en la agregación de SNCA. Se ha sugerido que el splicing alternativo afecta a la agregación de SNCA. Una deleción del exón 3 o 5 predice consecuencias funcionales: mientras que la deleción del exón 3 (SNCA126) conduce a la interrupción del dominio de interacción proteína-membrana N-terminal que puede conducir a una menor agregación, y la deleción del exón 5 (SNCA112) puede resultar en una mayor agregación debido a un acortamiento significativo del C-terminal no estructurado (Lee et al., 2001; Beyer, 2006). En el córtex frontal de los pacientes con DCL, SNCA112 está notablemente aumentado en comparación con los controles (Beyer et al., 2008), mientras que los niveles de SNCA126 están disminuidos en el córtex prefrontal de los pacientes con DCL (Beyer et al., 2006). Por el contrario, la expresión de SNCA126 mostró un aumento en la corteza frontal de los cerebros con EP y no hubo diferencias significativas en la MSA (Beyer et al., 2008). SNCA98 es una variante de empalme específica del cerebro que carece de los exones 3 y 5 y presenta diferentes niveles de expresión en varias áreas del cerebro fetal y adulto. Se ha informado de la sobreexpresión de SNCA98 en las cortezas frontales de DCL, EP (Beyer et al., 2007) y MSA (Beyer et al., 2008) en comparación con los controles. Además, se ha informado de que el proceso post-transcripcional que da lugar al uso alternativo del 3′UTR tiene efectos sobre la estabilidad y la localización del ARNm (Fabian et al., 2010; Rhinn et al., 2012; Yeh y Yong, 2016). Se justifica una mayor investigación sobre la propensión a la agregación de las diferentes isoformas conocidas de la proteína SNCA y la composición de los cuerpos de Lewy. Además, nuestro estudio sentó las bases para los análisis de cuantificación de ARNm de los transcritos previamente conocidos y nuevos en una muestra de mayor tamaño compuesta por sujetos con una gama de etapas clinicopatológicas utilizando varias regiones del cerebro de cada sujeto. Estos análisis del paisaje transcriptómico específico de la región cerebral de SNCA en el contexto de la gravedad neuropatológica serán informativos con respecto al papel de las isoformas de transcripción específicas de SNCA en la progresión de las etapas neuropatológicas y la gravedad de los cuerpos de Lewy y la densidad de las neuritas de Lewy.
En este trabajo, nos centramos en la creación de un estándar de secuenciación y análisis para analizar los datos de gDNA y cDNA dirigidos generados a partir de los mismos sujetos. Se trata de un potente enfoque que permite potencialmente el escalonamiento de las secuencias de ADNg en la región completa de un gen concreto basándose en la heterocigosidad en la secuencia de las isoformas de transcripción de longitud completa. Los datos de gDNA dirigidos por PacBio en este estudio produjeron bloques desfasados que cubrían el 81% de la región de 114 kb centrada en SNCA, con el bloque desfasado más largo superando los 54 kb. Como el desfase del ADNg está limitado por la longitud de la lectura y la heterocigosidad, el aumento de la longitud de la lectura probablemente generará bloques de fase más grandes.
El análisis de variantes del ADNg confirmó las repeticiones cortas en tándem (STRs) conocidas e identificadas en las regiones intrónicas. Por ejemplo, anteriormente, utilizando la secuenciación en fase por clonación y la secuenciación Sanger, descubrimos cuatro haplotipos distintos dentro de una región intrónica rica en CT que comprendía un grupo de secuencias repetitivas variables (Lutz et al., 2015). Demostramos que un haplotipo específico, denominado haplotipo 3, confería el riesgo de desarrollar la patología de los cuerpos de Lewy en los pacientes de Alzheimer. Aquí, validamos la secuencia de esta región altamente polimórfica de baja complejidad y sus cuatro haplotipos definidos. Aunque el tamaño de nuestra muestra era pequeño, el “haplotipo 3” estaba presente exclusivamente en los pacientes con la enfermedad (un paciente con EP y dos con DCL), lo que concuerda con nuestros hallazgos anteriores. Los resultados piloto y nuestra publicación anterior proporcionan la premisa para repetir los análisis de asociación de sinucleinopatías con STRs y haplotipos estructurales definidos con precisión, es decir, por lecturas largas, utilizando un tamaño de muestra mayor.
Nuestro trabajo demostró la capacidad del sistema PacBio Sequel para descubrir nuevos transcritos de longitud completa y caracterizar el repertorio completo de transcripciones de longitud completa de un gen implicado en una enfermedad. Además, también demostramos que las lecturas largas de ADNg definen con mayor precisión las variantes estructurales cortas y los haplotipos, incluidos los STR, y por ello pueden facilitar el descubrimiento y la validación de variantes asociadas a enfermedades distintas de los SNP. En conjunto, este nuevo conocimiento es muy valioso y aplicable en el avance de nuestra comprensión de las etiologías genéticas, que pueden implicar perturbaciones en el paisaje de la transcripción, que subyace a las enfermedades humanas complejas, incluyendo los trastornos neurodegenerativos relacionados con la edad, tales como sinucleinopatías.
Disponibilidad de datos
Las tres células SMRT de los datos brutos de ADNg están disponibles en Zenodo.org con doi: 10.5281/zenodo.1560688. La celda SMRT de datos brutos de ADNc está disponible en Zenodo.org con la dirección: 10.5281/zenodo.1581809. Los resultados procesados de gDNA y cDNA, incluyendo variantes de gDNA e isoformas de cDNA, están disponibles en Zenodo.org con doi: 10.5281/zenodo.3261805.
Contribuciones de los autores
OC-F contribuyó a la concepción y diseño del estudio. ET y WR organizaron las bases de datos de secuencias, realizaron los análisis de secuenciación y prepararon todas las figuras y tablas. O-CG y JB se encargaron de los tejidos cerebrales y de la preparación de las muestras nucleicas. TH generó los conjuntos de datos de secuenciación. SK diseñó y obtuvo los reactivos. OC-F, ET y WR redactaron el primer borrador del manuscrito. OC-F obtuvo la financiación. Todos los autores contribuyeron a la preparación del manuscrito, leyeron y aprobaron la versión presentada.
Financiación
Este trabajo fue financiado en parte por los Institutos Nacionales de Salud/Instituto Nacional de Trastornos Neurológicos y Accidentes Cerebrovasculares (NIH/NINDS).
Declaración de conflicto de intereses
ET, WR, TH y SK son o fueron empleados de Pacific Biosciences en el momento del estudio.
Los autores restantes declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un posible conflicto de intereses.
Agradecimientos
Este manuscrito se ha publicado como una preimpresión en BioRxiv (Tseng et al, 2019). https://www.biorxiv.org/content/10.1101/524827v1.
Material complementario
El material complementario de este artículo se puede encontrar en línea en: https://www.frontiersin.org/articles/10.3389/fgene.2019.00584/full#supplementary-material
Beach, T. G., Adler, C. H., Sue, L. I., Serrano, G., Shill, H. A., Walker, D. G., et al. (2015). Estudio de Arizona sobre el envejecimiento y los trastornos neurodegenerativos y programa de donación de cerebro y cuerpo. Neuropathology 35, 354-389. doi: 10.1111/neup.12189
PubMed Abstract | CrossRef Full Text | Google Scholar
Beyer, K. (2006). La estructura de la Α-sinucleína, la modificación postraduccional y el splicing alternativo como potenciadores de la agregación. Acta Neuropathol. 112, 237-251. doi: 10.1007/s00401-006-0104-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Beyer, K., and Ariza, A. (2012). La modificación postraduccional de la alfa-sinucleína y el splicing alternativo como desencadenantes de la neurodegeneración. Mol. Neurobiol. 47, 509-524. doi: 10.1007/s12035-012-8330-5
CrossRef Full Text | Google Scholar
Beyer, K., Domingo-Sàbat, M., Humbert, J., Carrato, C., Ferrer, I., and Ariza, A. (2008). Expresión diferencial de las isoformas de alfa-sinucleína, parkina y sinfilina-1 en la enfermedad de los cuerpos de Lewy. Neurogenetics 9, 163-172. doi: 10.1007/s10048-008-0124-6
PubMed Abstract | CrossRef Full Text | Google Scholar
Beyer, K., Domingo-Sàbat, M., Lao, J. I., Carrato, C., Ferrer, I., and Ariza, A. (2007). Identificación y caracterización de una nueva isoforma de alfa-sinucleína y su papel en las enfermedades de los cuerpos de Lewy. Neurogenetics 9, 15-23. doi: 10.1007/s10048-007-0106-0
CrossRef Full Text | Google Scholar
Beyer, K., Humbert, J., Ferrer, A., Lao, J. I., Carrato, C., pez, D. L., et al. (2006). Niveles bajos de ARNm de alfa-sinucleína 126 en la demencia con cuerpos de Lewy y la enfermedad de Alzheimer. Neuroreport 17, 1327-1330. doi: 10.1097/01.wnr.0000224773.66904.e7
PubMed Abstract | CrossRef Full Text | Google Scholar
Fabian, M. R., Sonenberg, N., y Filipowicz, W. (2010). Regulation of mRNA translation and stability by microRNAs. Annu. Rev. Biochem. 79, 351-379. doi: 10.1146/annurev-biochem-060308-103103
PubMed Abstract | CrossRef Full Text | Google Scholar
Edgar, R. C. (2004). MUSCLE: alineación de secuencias múltiples con alta precisión y alto rendimiento. Nucleic Acids Res. 32, 1792-1797. doi: 10.1093/nar/gkh340
PubMed Abstract | CrossRef Full Text | Google Scholar
Kalivendi, S. V., Yedlapudi, D., Hillard, C. J., and Kalyanaraman, B. (2010). Los oxidantes inducen el empalme alternativo de Α-sinucleína: implicaciones para la enfermedad de Parkinson. Free Radic. Biol. Med. 48, 377-383. doi: 10.1016/j.freeradbiomed.2009.10.045
PubMed Abstract | CrossRef Full Text | Google Scholar
Kim, W. S., Gedal, K. K., y Halliday, G. M. (2014). Biología de la alfa-sinucleína en las enfermedades de los cuerpos de Lewy. Alzheimers Res. Ther. 6, 1-9. doi: 10.1186/s13195-014-0073-2
CrossRef Full Text | Google Scholar
Kohli, M. (2017). La variante del receptor de andrógenos AR-V9 se coexpresa con AR-V7 en las metástasis del cáncer de próstata y predice la resistencia a la abiraterona. Clin. Cancer Res. 23, 1-13. doi: 10.1158/1078-0432.CCR-17-0017
CrossRef Full Text | Google Scholar
Lee, H.-J., Choi, C., y Lee, S. J. (2001). La Α-sinucleína unida a la membrana tiene una alta propensión a la agregación y la capacidad de sembrar la agregación de la forma citosólica. J. Biol. Chem. 277, 671-678. doi: 10.1074/jbc.M107045200
CrossRef Full Text | Google Scholar
Lutz, M. W., Saul, R., Linnertz, C., Glenn, O.-C., Roses, A. D., and Chiba-Falek, O. (2015). Un haplotipo rico en citosina-timina (CT) en el intrón 4 de SNCA confiere riesgo de patología de cuerpos de Lewy en la enfermedad de Alzheimer y afecta a la expresión de SNCA. Alzheimers Dement. 11, 1133-1143. doi: 10.1016/j.jalz.2015.05.011
PubMed Abstract | CrossRef Full Text | Google Scholar
Martin, M., Patterson, M., Garg, S., Fischer, S. O., Pisanti, N., Klau, G. W., et al. (2016). WhatsHap: fast and accurate read-based phasing. bioRxiv . doi: 10.1101/085050
CrossRef Full Text | Google Scholar
McKeith, I. G., Dickson, D. W., Lowe, J., Emre, M., O’Brien, J. T., Feldman, H., et al. (2005). Diagnosis and management of dementia with Lewy bodies: third report of the DLB consortium. Neurology 65, 1863-1872. doi: 10.1212/01.wnl.0000187889.17253.b1
PubMed Abstract | CrossRef Full Text | Google Scholar
McKeith, I. G., Perry, E. K., y Perry, R. H. (1999). Report of the second dementia with Lewy body international workshop: diagnosis and treatment. Consorcio sobre la demencia con cuerpos de Lewy. Neurology 53, 902-905. doi: 10.1212/WNL.53.5.902
PubMed Abstract | CrossRef Full Text | Google Scholar
McLean, J. R., Hallett, P. J., Cooper, O., Stanley, M., and Isacson, O. (2012). Niveles de expresión de transcripción de la alfa-sinucleína de longitud completa y sus tres variantes empalmadas alternativamente en las regiones del cerebro de la enfermedad de Parkinson y en un modelo de ratón transgénico de sobreexpresión de alfa-sinucleína. Mol. Cell. Neurosci. 49, 230-239. doi: 10.1016/j.mcn.2011.11.006
PubMed Abstract | CrossRef Full Text | Google Scholar
Poplin, R., Ruano-Rubio, V., DePristo, M. A., Fennell, T. J., Carneiro, M. O., Van der Auwera, G. A., et al. (2018). Scaling accurate genetic variant discovery to tens of thousands of samples. bioRxiv . doi: 10.1101/201178
CrossRef Full Text | Google Scholar
Rhinn, H., Qiang, L., Yamashita, T., Rhee, D., Zolin, A., Vanti, W., et al. (2012). Uso alternativo de la transcripción de Α-sinucleína como un mecanismo convergente en la patología de la enfermedad de Parkinson. Nat. Commun. 3, 889-821. doi: 10.1038/ncomms2032
CrossRef Full Text | Google Scholar
Sandor, C., Robertson, P., Lang, C., Heger, A., Booth, H., Vowles, J., et al. (2017). El perfil transcriptómico de las neuronas dopaminérgicas purificadas derivadas de pacientes identifica perturbaciones convergentes y terapéuticas para la enfermedad de Parkinson. Hum. Mol. Genet. 54, ddw412-ddw415. doi: 10.1093/hmg/ddw412
CrossRef Full Text | Google Scholar
Treutlein, B., Gokce, O., Quake, S. R., and Südhof, T. C. (2014). Cartografía del splicing alternativo de la neurexina mapeado por secuenciación de ARNm de una sola molécula de lectura larga. Proc. Acad. Sci. 111, E1291-E1299. doi: 10.1073/pnas.1403244111
PubMed Abstract | CrossRef Full Text | Google Scholar
Tseng, E., Rowell, W. J., Omolara-Chinue, G., Hon, T., Barrera, J., Kujawa, S., et al. (2019). The landscape of SNCA transcripts across synucleinopathies: new insights from long reads sequencing analysis. bioRxiv . doi: 10.1101/524827
CrossRef Full Text | Google Scholar
Tseng, E., Tang, H.-T., AlOlaby, R. R., Hickey, L., and Tassone, F. (2017). Expresión alterada del paisaje de variantes de empalme de FMR1 en portadores de premutación. Biochim. Biophys. Acta Gene. Regul. Mech. 1860, 1117-1126. doi: 10.1016/j.bbagrm.2017.08.007
CrossRef Full Text | Google Scholar
Xu, W., Tan, L., y Yu, J.-T. (2014). El vínculo entre el gen SNCA y el parkinsonismo. Neurobiol. Aging 36, 1-14. doi: 10.1016/j.neurobiolaging.2014.10.042
CrossRef Full Text | Google Scholar
Yeh, H.-S., y Yong, J. (2016). Poliadenilación alternativa de los ARNm: La región 3′-no traducida importa en la expresión génica. Mol. Cell 39, 281-285. doi: 10.14348/molcells.2016.0035
PubMed Abstract | CrossRef Full Text | Google Scholar