Preface
Questo articolo offre una visione personale di ciò che potrebbe essere necessario affinché la terapia fagica possa finalmente sfondare come strumento antibatterico mainstream. È influenzato dai fallimenti e dalle incertezze storiche e recenti nel campo della terapia fagica e mira a trovare soluzioni basate su tecnologie future ed emergenti che dovrebbero modellare la scienza e la società di domani.
Terapia fagica
I batteriofagi (fagi) sono i virus dei batteri. Da tempo immemorabile controllano la crescita e la diffusione dei loro ospiti batterici. I virus batterici sono le entità vitali più onnipresenti nella nostra biosfera. Si stima che ci siano 10 milioni di volte più virus negli oceani di quante siano le stelle nell’universo e se tutti i fagi sulla Terra fossero impilati uno sull’altro, questa torre si estenderebbe più lontano delle 60 galassie più vicine (Suttle, 2013). Possono essere facilmente trovati ovunque i batteri prosperino: nelle fogne, nei fiumi o nelle urine e nelle feci dei pazienti. I fagi dei patogeni batterici umani sono più spesso composti da una testa icosaedrica, una sfera con 20 facce piane fatta di proteine e contenente un genoma di acido nucleico, alla quale è attaccata una coda proteica. Quando un fago strettamente litico aderisce con le sue fibre e punte della coda alla superficie del suo batterio bersaglio, la guaina della coda simile a una siringa si contrae e il nucleo della coda viene spinto attraverso la parete cellulare batterica, iniettando il genoma del fago nel periplasma della cellula batterica. Immediatamente, il DNA batterico e il macchinario di sintesi proteica vengono dirottati per costruire copie del fago. Alcuni fagi tagliano il DNA batterico in pezzi. Dopo un periodo di latenza da minuti a ore, i fagi appena formati scoppiano dai loro ospiti batterici, che vengono uccisi nel processo. La progenie del fago, che può arrivare a centinaia per batterio, va a cercare nuovi batteri ospiti da infettare. Come tali, i fagi possono essere considerati come antimicrobici auto-replicanti. È importante notare che i fagi si sono evoluti per infettare solo determinati batteri bersaglio e sono innocui per le cellule dei mammiferi.
Le prime prove di agenti simili ai virus con attività antibatterica furono riportate dal batteriologo inglese Frederick Twort e dal microbiologo franco-canadese Felix d’Hérelle rispettivamente nel 1915 e nel 1917 (Sulakvelidze et al., 2001). Nel 1919, d’Hérelle sfruttò per la prima volta il potenziale terapeutico dei fagi quando li usò per curare un ragazzo che soffriva di dissenteria a Parigi. La terapia fagica fu immediatamente riconosciuta come un approccio terapeutico per trattare le infezioni batteriche e la commercializzazione dei preparati per la terapia fagica fu intrapresa da diverse aziende, come L’Oréal in Europa e Eli Lilly Company negli Stati Uniti (Sulakvelidze et al., 2001). Nel 1923, il microbiologo georgiano Giorgi Eliava fondò l’Istituto Eliava a Tbilisi, Georgia, dedicato alla ricerca sulla terapia fagica. Fu l’inizio della ricerca e dello sviluppo della terapia fagica nell’ex Unione Sovietica. Tuttavia, i primi usi della terapia fagica erano spesso inaffidabili e la ricerca sugli antibiotici era anche in corso. Il successo dell’uso della penicillina durante la seconda guerra mondiale e la sua successiva commercializzazione a livello mondiale portò gli scienziati occidentali a perdere interesse nella terapia fagica. I ricercatori sovietici, al contrario, continuarono a sviluppare la terapia fagica e a pubblicare i loro risultati, ma a causa della cortina di ferro la loro conoscenza ed esperienza non si diffuse nel mondo (Sulakvelidze et al., 2001). All’alba del terzo millennio, il crescente peso sanitario delle infezioni da batteri resistenti agli antibiotici (Cassini et al., 2019) ha suscitato un rinnovato interesse a livello mondiale per la terapia fagica come un valido strumento aggiuntivo per la gestione clinica delle infezioni batteriche (Thiel, 2004). In tutto il mondo stanno nascendo centri di terapia fagica, sulle orme dell’Istituto Eliava e dell’unità di terapia fagica dell’Istituto Hirszfeld di Wrocław, in Polonia (Miêdzybrodzki et al, 2012).
L’anno 2035
Passiamo alla futura Terra del 2035, un mondo cupo caratterizzato da sovrappopolazione umana, grandi sconvolgimenti dell’ecosistema, riscaldamento globale e xenofobia.
Mentre era a mollo nel suo bagno, il dottor John Iverian, un microbiologo in pensione, ha improvvisamente sentito una puntura estremamente dolorosa dietro il collo, seguita da un suono simile all’elica di un piccolo aereo. Ha schermato l’ambiente e con la coda dell’occhio ha visto uno strano insetto di grandi dimensioni con lunghe zampe e antenne inquietanti seduto sul muro accanto alla sua vasca da bagno di design. Osuri, il sistema di gestione domestica del loft di Iverian nel centro di Anversa, identificò l’insetto come la cimice marrone marmorizzata Halyomorpha halys, che si era diffusa in tutto il mondo. La relazione di Osuri, proiettata su uno degli schermi video del bagno, ha menzionato che le persone che sono state morse, inizialmente hanno sperimentato una piccola piaga rossa nella zona del morso della loro pelle. Se lasciata incustodita, la ferita del morso si gonfiava e produceva pus. Stanco e confuso, Iverian, noncurante, uscì dal bagno e andò a letto. Aveva deciso di non eseguire l’elaborata procedura di trattamento della ferita, che era stata fortemente consigliata da Osuri. La mattina dopo, tuttavia, il morso si era trasformato in una ferita necrotica che mostrava chiari segni di infezione.
Ansiosamente, Iverian attivò il suo dispositivo Phage-BEAM. BEAM stava per “Bedside Energized Anti-Microbial”. Il dispositivo aveva le dimensioni e la forma di una scatola da scarpe, ma con un aspetto più elegante e raffinato. Il nome del dispositivo e il suo produttore erano disegnati in lettere colorate sul lato della scatola smaltata bianca senza cuciture. Iverian estrasse un tampone dalla sua confezione sterile e lo passò delicatamente su tutta l’area della ferita, assicurandosi che l’essudato della ferita bagnasse completamente la punta di cotone del tampone. Quando il tampone si avvicinò all’area “inserisci campione” della scatola, una piccola porta si aprì come per magia, liberando un ologramma alto 10 pollici di un tecnico di laboratorio, chiamato Marcia. Lei mostrò a Iverian dove agganciare il campione. Marcia è stata sviluppata per guidare i clienti attraverso la procedura del test. “Per i migliori risultati, per favore inserisca una nuova cartuccia di bioinchiostro fagico, dottor Iverian”, disse Marcia. Proprio come accadeva per le stampanti 2D di un tempo, il costo delle cartucce di bio-inchiostro era quasi pari al costo del dispositivo Phage-BEAM stesso. Secondo “Business Insider”, il bio-inchiostro fagico era il secondo liquido più costoso sulla Terra, dietro Chanel No. 8. Fortunatamente, come uno degli inventori, Iverian aveva ottenuto il diritto di avere sempre la versione più recente di questo dispositivo a sua disposizione, compresa una fornitura continua di reagenti, gratuitamente.
Iverian sapeva perfettamente come funzionava il dispositivo, quindi non aveva bisogno dell’aiuto di Marcia. Per prima cosa, il DNA veniva estratto dalla punta del tampone e il metagenoma – tutto il materiale genetico presente nel campione, compresi i batteri infettanti – veniva determinato. Successivamente, questi dati genetici sono stati inviati a un server protetto “Phage XChange” dove un complesso algoritmo guidato dall’intelligenza artificiale ha predetto la sequenza del genoma del fago che più probabilmente avrebbe liso i batteri infettanti identificati nel metagenoma e che avrebbe dovuto suscitare la reazione immunitaria più debole nel paziente. I dati del genoma del fago sono stati inviati al dispositivo Phage-BEAM, che ha sintetizzato prima il genoma del fago e poi il fago, utilizzando un sistema proprietario di produzione di fagi senza batterio.
In 1 ora dopo il campionamento, il dispositivo avrebbe prodotto un prodotto fagico terapeutico pronto all’uso. I risultati della procedura passo dopo passo sarebbero stati trasmessi all’enorme schermo video nel salotto di Iverian. Seduto nella sua poltrona LC2, ascoltando la Grande Messa in Do minore di Mozart, Iverian aspettava con ansia i risultati. Aveva un brutto presentimento. Il risultato mandò dei brividi lungo la schiena di Iverian. Patogeno batterico identificato: Streptococcus pyogenes ceppo FE-2033! Osuri attivò immediatamente il protocollo di allarme per le infezioni, inviando un messaggio al Centro Mondiale per il Controllo delle Malattie e proiettando preoccupanti informazioni di base sul ceppo batterico letale mangia-carne, che era considerato una minaccia imminente per la salute pubblica dal 2033. Per un momento, Iverian prese in considerazione l’idea di asportare la ferita infetta e alcuni tessuti sani circostanti con un coltello da cucina, ma si calmò e decise di aspettare e applicare l’imminente prodotto Phage-BEAM. Un’ora dopo, il dispositivo Phage-BEAM aveva prodotto dei fagi sintetici. Questi fagi sono stati poi mescolati con i batteri isolati, in un modulo di convalida, per testare la loro efficacia in vitro. Quindici minuti dopo, Iverian ha avuto il via libera per iniziare il trattamento. Iverian ha applicato i fagi in una medicazione per ferite a lento rilascio a base di idrogel, che era stata prima mescolata con la sospensione fagica concentrata prodotta dal dispositivo Phage-BEAM, e conteneva anche antibiotici sinergici. L’idrogel ha temporaneamente alleviato il dolore, il che lo ha ulteriormente calmato. Iverian ha ripetuto l’applicazione dell’idrogel carico di fagi e antibiotici una volta al giorno. L’infezione della ferita è migliorata entro 24 ore e dopo 7 giorni la ferita era quasi completamente guarita. L’infezione potenzialmente pericolosa per la vita di Iverian è stata trattata con successo, tempestivamente e senza lasciare la sua casa. Ma, per molti decenni precedenti, non era certo che la terapia fagica avrebbe sfondato per diventare uno strumento antibatterico ampiamente applicato e clinicamente utile. Il mondo medico ci aveva messo un po’ a capire che la terapia fagica non doveva essere identica alla terapia antibiotica, e questo principalmente a causa delle peculiarità degli agenti attivi, i fagi.
Alcune rilevanti peculiarità dei fagi
I fagi presentano una serie di proprietà che differiscono dagli antibiotici e hanno ostacolato il loro sviluppo come prodotti farmaceutici e l’applicazione nella terapia. In primo luogo, tendono ad essere specifici su quali batteri infettano. Nel migliore dei casi colpiscono una parte considerevole di una singola specie batterica, ma nel peggiore dei casi infettano solo un piccolo numero di ceppi all’interno di una specie. I fagi terapeutici possono quindi essere selezionati per uccidere principalmente una specie batterica, o un suo sottogruppo clinicamente rilevante, e risparmiare i batteri benefici del paziente (ad esempio, l’intestino, la pelle o la flora commensale orale). La maggior parte degli antibiotici utilizzati di routine, al contrario, hanno un ampio spettro di attività, che può causare “danni collaterali” ai microbiomi commensali del paziente, che a sua volta può provocare effetti avversi come la selezione di specie batteriche resistenti agli antibiotici (ad esempio, Clostridium difficile) o diarrea associata agli antibiotici (Jernberg et al., 2010). Lo svantaggio della specificità fagica è che i batteri infettanti devono essere identificati prima di iniziare la terapia fagica. Nella terapia antibiotica empirica, al contrario, vengono tipicamente utilizzati cocktail di antibiotici ad ampio spettro che colpiscono una moltitudine di batteri Gram-positivi e Gram-negativi, e diversi funghi. Quando si conoscono più informazioni (per esempio, dalla coltura batterica), il trattamento può consistere in antibiotici a spettro stretto, che mirano più specificamente ai batteri o ai funghi identificati come causa della malattia.
In secondo luogo, batteri e fagi sono coinvolti in una relazione ospite-parassita. I fagi strettamente litici sono onnipresenti nell’ambiente e richiedono la morte del loro ospite batterico per completare il loro ciclo vitale. Senza ospiti, i fagi non possono esistere. I fagi impongono la selezione per ospiti resistenti, che a loro volta impongono la selezione per fagi efficaci. Questo si traduce in quella che viene chiamata “coevoluzione antagonistica”, una corsa agli armamenti tra batteri e fagi, caratterizzata dall’evoluzione reciproca della resistenza batterica e dell’infettività dei fagi (Buckling e Rainey, 2002). Proprio come con la maggior parte degli antimicrobici, i batteri diventeranno quindi anche resistenti ai fagi (Luria e Delbrück, 1943; Schooley et al., 2017), ma, a differenza degli antibiotici statici, i fagi hanno la capacità di superare la resistenza batterica (Buckling e Rainey, 2002). Ci sono tuttavia indicazioni che batteri e fagi non aumenteranno indefinitamente la loro rispettiva resistenza e infettività (Fortuna et al., 2019).
Approcci di fagoterapia
Al momento del revival della terapia fagica nei primi anni 2000, erano stati sviluppati due approcci distinti di terapia fagica (Pirnay et al., 2011). In quello che potrebbe essere chiamato l’approccio “one-size-fits-all”, sono stati applicati cocktail di fagi ad ampio spettro definiti, che avrebbero dovuto colpire la maggior parte dei batteri sospettati di causare determinate malattie infettive. Questi cocktail di fagi ad ampio spettro predefiniti sono stati sviluppati, prodotti e testati all’interno degli attuali modelli farmaco-economici, che erano stati progettati per soddisfare i farmaci “statici” come gli antibiotici. Tuttavia, i cocktail di fagi veramente ad ampio spettro, attivi contro la maggior parte dei batteri Gram-positivi e/o Gram-negativi comunemente incontrati nelle malattie infettive dovevano contenere grandi quantità di fagi e si sono rivelati molto difficili da sviluppare. Era possibile sviluppare cocktail di fagi a spettro più ristretto, attivi contro una o poche specie batteriche, da utilizzare in determinate indicazioni e tenendo presente che le specie batteriche infettanti erano note in anticipo. Per alcune specie batteriche, come lo Staphylococcus aureus, sono stati isolati e caratterizzati fagi che mostrano un range di ospiti eccezionalmente ampio (Vandersteegen et al., 2011). In PhagoBurn, uno studio controllato randomizzato, il successo nell’ottenere una riduzione sostenuta dei carichi di Pseudomonas aeruginosa nelle ferite da ustione era legato alla suscettibilità iniziale al cocktail di fagi (Jault et al., 2019). Tuttavia, un terzo dei pazienti inclusi ha dimostrato di ospitare preesistenti ceppi di P. aeruginosa resistenti al cocktail di fagi, che consisteva di non meno di 12 fagi litici di P. aeruginosa. Inoltre, i cocktail di fagi inizialmente progettati per essere efficaci dovevano essere regolarmente aggiornati (ad esempio, integrati con nuovi fagi) in risposta all’emergere della resistenza dei fagi o al coinvolgimento di nuovi ceppi clinicamente rilevanti in circolazione. Infine, non si sapeva se affrontare i batteri con alte concentrazioni di cocktail di fagi fissi avrebbe causato l’emergere, la diffusione e la persistenza della resistenza dei fagi batterici negli ospedali e nell’ambiente, in modo simile a quanto era successo con l’uso massiccio di antibiotici.
Nei concetti di terapia fagica personalizzata, uno o più fagi sono stati selezionati da una banca di fagi, o dall’ambiente, ed eventualmente adattati (selezione in vitro di mutanti fagici che mostrano una maggiore infettività) per infettare in modo più efficiente i batteri isolati dal sito di infezione del paziente (Friman et al, 2016). Alcuni centri di terapia fagica hanno istituito e mantenuto grandi banche di fagi terapeutici, che venivano regolarmente aggiornati con nuovi fagi, ampliando e adattando la gamma di ospiti della banca alle popolazioni batteriche in continua evoluzione. Gli approcci terapeutici fagici personalizzati erano potenzialmente più sostenibili, in quanto solo il batterio infettante viene preso di mira, con conseguente minore pressione di selezione verso la resistenza dei fagi batterici. Tuttavia, erano anche più elaborati e logisticamente complessi degli approcci a taglia unica, con ceppi batterici e fagi corrispondenti inviati in tutto il mondo (Figura 1). Inoltre, i concetti di medicina di precisione non erano, in generale, compatibili con la maggior parte dei percorsi di sviluppo e licenza di prodotti medicinali (farmaci negli Stati Uniti), che richiedevano diversi anni e milioni di euro (dollari) per essere completati, e questo per ogni fago nella banca (Verbeken et al., 2012).
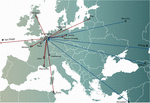
Figura 1. Trasferimenti internazionali di fagi da (frecce rosse) e verso (frecce blu) il Queen Astrid Military Hospital (QAMH) di Bruxelles in vista di applicazioni cliniche nel periodo 2015-2020. A livello nazionale, i fagi sono stati spediti dal QAMH a cinque ospedali universitari (non mostrato). Inoltre, la selezione dei fagi corrispondenti spesso comprendeva il trasferimento degli isolati batterici dei pazienti, e cinque pazienti internazionali (due dalla Francia, due dai Paesi Bassi e uno dalla Tunisia) sono stati trasferiti a Bruxelles per la terapia con i fagi.
Entrare nella biologia sintetica
Con l’inizio del terzo millennio, gli approcci della biologia sintetica sono stati sempre più sviluppati per ridurre la specificità dei fagi e l’emergere della resistenza dei fagi batterici (ad es, structure-guided design) (Pires et al., 2016; Dunne et al., 2019). Ad esempio, sono state elaborate piattaforme basate sul lievito per la commutazione della proteina della coda del fago per ingegnerizzare fagi ibridi con una gamma di ospiti più prevedibile ed estesa (Ando et al., 2015; Yosef et al., 2017) e strategie di ingegneria genetica (ad es, strumenti di editing CRISPR-Cas) sono state sviluppate per affrontare altri aspetti come le interazioni negative paziente-fago (ad esempio, la risposta immunitaria anti-fago) (Brown et al., 2017), la potenziale emergenza e diffusione di meccanismi di resistenza dei fagi batterici e il rilascio di contenuti batterici dannosi come le endotossine (Hwang et al., 2018). I genomi fagici sintetici dovevano essere riavviati per produrre prole fagica (Barbu et al., 2016; Pires et al., 2016), attraverso la trasformazione delle forme L di Escherichia coli o Listeria monocytogenes (Kilcher et al., 2018), o utilizzando sistemi di trascrizione-traslazione senza cellule (TXTL) (Rustad et al., 2018). I quadri normativi occidentali avevano gradualmente iniziato a soddisfare gli approcci di fagoterapia di precisione e personalizzati utilizzando fagi naturali (Pirnay et al., 2018), fagi ingegnerizzati (Dedrick et al., 2019) e fagi sintetici.
Lo sviluppo di dispositivi di produzione di fagi terapeutici ad hoc e in loco, come Phage-BEAM, non è andato liscio, ha ricordato Iverian. Per cominciare, è stato necessario l’abbinamento e la progettazione di fagi in silico basati sull’intelligenza artificiale (AI). L’apprendimento profondo (Martorell-Marugán et al., 2019), un sottoinsieme del Machine Learning, è stato scelto per cercare collegamenti tra i genomi batterici e i genomi dei fagi infettanti, perché era più facile da scalare a un numero maggiore di campioni. Ad esempio, i metodi di apprendimento profondo non hanno richiesto la cosiddetta estrazione di caratteristiche, che richiederebbe l’annotazione a livello di geni/proteine dei genomi fagici e batterici e limiterebbe le previsioni ad alcune relazioni note tra caratteristiche batteriche e fagiche, come le strutture delle fibre della coda del fago che si legano a specifici recettori della parete cellulare batterica. Come lato negativo, doveva essere alimentato da una fornitura continua di massicce quantità di dati, collegando i genomi dei fagi litici ai genomi dei batteri ospiti, ed è qui che la scarpa si è bloccata. Il sequenziamento del genoma intero si era lentamente percolato nella pratica della microbiologia clinica (American Academy of Microbiology, 2016), ma gli istituti di ricerca e le aziende farmaceutiche non erano propensi a sottoporre i loro dati a un unico database centralizzato, e non si trovavano investitori disposti e capaci di acquisire i dati disponibili e/o di produrre quantità sufficienti di nuovi dati. Un secondo ostacolo da superare era l’indisponibilità di una sintesi rapida, affidabile ed economica di grandi molecole di DNA. Le tecniche iniziali di sintesi del DNA erano basate sulla chimica organica e producevano molecole di DNA relativamente piccole. La sintesi de novo dei genomi fagici richiedeva l’assemblaggio di diversi frammenti di genoma (Barbu et al., 2016; Pires et al., 2016; Lemire et al., 2018) nel lievito Saccharomyces cerevisiae, utilizzando cromosomi artificiali di lievito (Ando et al., 2015), o l’assemblaggio chimico (Gibson et al., 2009). Lo sviluppo di una nuova tecnica per sintetizzare il DNA, basata sugli enzimi di sintesi del DNA che si trovano nelle cellule del sistema immunitario (Palluk et al., 2018), ha facilitato la sintesi del genoma dei fagi. Infine, è stato necessario superare alcuni ostacoli per sviluppare sistemi generici di produzione di fagi senza cellule in grado di produrre fagi ad alti titoli ed esibire gli stessi livelli di infettività batterica dei loro analoghi naturali.
La svolta
Il problema principale è stato che si è rivelato molto difficile raccogliere le massicce quantità di sequenze di fagi e genomi batterici collegati, necessarie per gli algoritmi AI di deep learning per prevedere e/o progettare sequenze di fagi con un livello di precisione terapeuticamente accettabile. Iverian ha ricordato che la vera svolta è avvenuta quando l’organizzazione no-profit “Phage XChange” ha lanciato la sua omonima piattaforma globale di governance dei fagi per creare una catena di approvvigionamento di fagi efficiente, standardizzata, sostenibile ed etica (Figura 2). Phage XChange consisteva principalmente in un modulo AI e un Distributed Ledger (DL) (Thiebes et al., 2020). Il modulo AI della piattaforma ha analizzato i genomi di fagi e batteri collegati per prevedere e progettare fagi potenti per i clienti. Ha anche previsto quali patogeni batterici necessitavano dell’attenzione più urgente, sulla base dell’Internet of Things (IoT) e dei Big Data e delle informazioni fornite dalle organizzazioni internazionali di sanità pubblica, come l’Organizzazione mondiale della sanità (OMS) e i Centri nazionali per il controllo delle malattie. Questi dati hanno indirizzato il sistema verso l’isolamento e la caratterizzazione dei fagi più urgenti.
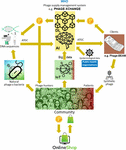
Figura 2. Visione di come potrebbe essere organizzata la catena di approvvigionamento dei fagi nel 2035. AI, intelligenza artificiale; ATGC, sequenza di DNA; BEAM, bedside energized anti-microbial; DL, distributed ledger; IoT, Internet of Things; P, PhageCoin; WHO, World Health Organization.
Il modulo DL della piattaforma ha garantito un input sufficiente, qualitativo e registrato di sequenze di fagi/genomi batterici collegati al modulo AI e idem la fornitura di sequenze di fagi ai clienti, in conformità alle disposizioni del protocollo di Nagoya (Expert round table on acceptance and re-implementation of bacteriophage therapy et al, 2018). Il DL registrava immutabilmente tutte le parti interessate (ad esempio, fornitori di dati, servizi di sequenziamento e clienti), le transazioni e i dettagli del contratto. Ha anche registrato l’esatta qualità, le specifiche e il peso del materiale fornito. Un algoritmo ha determinato la non ridondanza e ha stimato il peso (ad esempio, la virulenza e la gamma di ospiti dei fagi) e la desiderabilità del materiale inviato. I fagi che hanno come obiettivo i patogeni batterici emergenti erano ovviamente i più ricercati. La maggior parte dei problemi di brevetto erano ovviati. La DL ha agito come un libro mastro dei pagamenti per assicurare che tutte le parti fossero pagate tempestivamente ed equamente. Un certo numero di PhageCoin (la criptovaluta della piattaforma) sono stati attribuiti ai fornitori in relazione alla qualità, peso e desiderabilità del materiale fornito. I clienti che estraevano i risultati di predizione (sequenze di fagi) attraverso la DL pagavano una quantità di PhageCoin, proporzionale al valore stimato dei fagi. Questi PhageCoin sono stati usati per mantenere la DL, per assicurare un afflusso sufficiente e continuo di materiale, e per espandere i dati sulla virulenza dei fagi e sulla gamma di ospiti (abbinando i fagi ai batteri). Un’ulteriore iniezione di fondi e un incentivo a fornire materiale è stato trovato nei produttori e fornitori di tutti i tipi di beni. Con i PhageCoin guadagnati istantaneamente, i fornitori di fagi potevano comprare online tutti i tipi di prodotti a prezzi fortemente ridotti, dalle attrezzature di laboratorio e scolastiche agli articoli sportivi. Questi beni erano forniti attraverso la sponsorizzazione aziendale. Diverse aziende affermate sponsorizzarono PhageXchange in cambio di riduzioni fiscali, pubblicità e l’immagine di un marchio socialmente responsabile. Il peso del materiale fornito, e quindi anche il suo valore, erano inizialmente sottovalutati, ma venivano rivalutati a intervalli regolari (iterazione) e ai fornitori venivano attribuiti più PhageCoin quando garantito. Anche se utile dal momento in cui è stata introdotta, la piattaforma ha avuto davvero successo solo quando è stata messa sotto la protezione dell’OMS, in analogia al sistema mondiale di tracciabilità, trasparenza, vigilanza e sorveglianza dei prodotti medicinali di origine umana (Warwick et al., 2013). Un accordo formale tra Phage XChange e l’OMS ha aumentato la fiducia internazionale nella sostenibilità a lungo termine della piattaforma e nella protezione dallo sfruttamento commerciale non etico. La ricerca di potenti fagi terapeutici divenne presto uno sforzo comunitario volto a risolvere la crisi della resistenza agli antibiotici, con “cacciatori di fagi” indipendenti, scuole, gruppi di scout, villaggi sulle rive del Rio delle Amazzoni, ecc, che isolano e presentano i fagi a Phage XChange, in cambio di PhageCoin. A margine di questo, varie aziende e istituzioni hanno sviluppato attrezzature e servizi periferici, come kit di isolamento dei fagi e piattaforme di sequenziamento e sintesi dei fagi (ad esempio, il dispositivo Phage-BEAM). In previsione di questi dispositivi, sono state offerte soluzioni intermedie, per cui i fagi stessi sono stati ottenuti attraverso la DL.
Epilog
Questa visione sul futuro della terapia fagica fornisce un finale ottimistico alla crisi della resistenza agli antibiotici. La produzione ad hoc e in loco di fagi sintetici, collegata a un sistema di gestione dei fagi globale e comunitario, si è rivelata un’arma in più gradita e accessibile (per i sistemi di sicurezza sociale) nella lotta contro le infezioni batteriche resistenti agli antibiotici. Tuttavia, non era una pallottola magica; era un supplemento sinergico agli antimicrobici stabiliti. La produzione istantanea e senza cellule di fagi sintetici, progettati o meno, presentava notevoli vantaggi rispetto ai fagi naturali prodotti classicamente (in ospiti batterici):
(i) Non era necessario mantenere banche fisiche di fagi terapeutici e spedire gli isolati batterici del paziente e i corrispondenti fagi terapeutici in tutto il mondo.
(ii) I fagi sintetici contro i batteri che causano eminenti minacce alla salute pubblica, come l’epidemia di E. coli O104:H4 del 2011 in Germania (Merabishvili et al., 2012), o batteri (sospettati di essere) utilizzati per il bioterrorismo (Joñczyk-Matysiak et al, 2014) potrebbe essere prodotta tempestivamente in loco.
(iii) Fagi contro i batteri che causano malattie potenzialmente letali, per i quali non erano disponibili ceppi ospiti di produzione non letali e la cui propagazione utilizzato per richiedere biosicurezza livello 3 (bsl-3) precauzioni di bio-contenimento, potrebbe essere sintetizzato in condizioni bsl-1.
(iv) Quando non è stato possibile isolare i fagi dai siti di campionamento, per esempio, perché i ceppi ospiti batterici utilizzati nelle tecniche di isolamento non erano suscettibili ai fagi desiderati, le sequenze genomiche (previste) dei fagi, estratte dai dati metagenomici (Reyes et al, 2010; Amgarten et al., 2018), potrebbero essere utilizzate per produrre fagi sintetici.
(v) Le preparazioni di fagi sintetici non contenevano molecole (o quantità minori di) che potrebbero avere un impatto negativo sui pazienti (ad esempio, endotossine).
(vi) I dispositivi sono stati adattati per produrre fagi sintetici durante i viaggi spaziali estesi e la colonizzazione dello spazio (Taylor e Sommer, 2005).
Ci sono poche possibilità che queste previsioni si avverino. È probabilmente troppo miope pensare che uno sforzo basato sulla comunità, sostenuto da organizzazioni di salute pubblica e gestito da una piattaforma globale, sostenibile ed etica, possa essere il cuore di una soluzione all’attuale crisi mondiale della resistenza agli antibiotici. Alcune parti del sistema proposto, come la produzione senza cellule di fagi sintetici utilizzando un dispositivo sul letto, hanno una ragionevole possibilità di essere realizzate, mentre altri elementi, come la sponsorizzazione aziendale, rimarranno probabilmente limitati al regno della fantascienza. Potete dire che sono un sognatore, quindi sentitevi liberi di svegliarmi nel 2035 per confrontarmi con la realtà!
Contributi dell’autore
J-PP ha concepito la visione e redatto il manoscritto.
Finanziamento
I costi di pubblicazione sono stati coperti dalla “Société Scientifique du Service Médical Militaire – Wetenschappelijke Vereniging van de Militaire Medische Dienst”.
Conflitto di interessi
L’autore dichiara che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che potrebbero essere interpretate come un potenziale conflitto di interessi.
Il curatore ha dichiarato di essere stato coautore dell’autore.
Riconoscimenti
La visione personale, o sogno, sviluppato in questo manoscritto è il risultato delle interazioni con molti colleghi ricercatori negli ultimi 15 anni. È impossibile nominarli tutti, ma non sarebbe giusto prendere tutti i crediti da solo. Pertanto, ho deciso di riconoscere alcuni di loro qui (in ordine alfabetico), con il rischio – o meglio, la certezza – di dimenticare alcuni importanti influenzatori: Joana Azeredo, Nata Bakuradze, Bob Blasdel, Dimitri Boeckaerts, Angus Buckling, Yves Briers, Pieter-Jan Ceyssens, Nina Chanishvili, Laurent Debarbieux, Sarah Djebara, Dorien Dams, Daniel De Vos, Quirin Emslander, Alan Fauconnier, Ville Friman, Andrzej Górski, Téa Glonti, Nino Grdzelishvili, Serge Jennes, Elene Kakabadze, Betty Kutter, Rob Lavigne, Cédric Lood, Alice Maestri, Khatuna Makalatia, Maya Merabishvili, Tobi Nagel, Thomas Rose, Patrick Soentjens, Michiel Stock, Rüdiger Trojok, An Van den Bossche, Mario Vaneechoutte, Gilbert Verbeken e Kilian Vogele.
Accademia americana di microbiologia (2016). Applicazioni del sequenziamento microbico clinico di nuova generazione: Report on an American Academy of Microbiology Colloquium Held in Washington, DC, in April 2015. Washington, DC: American Society for Microbiology.
Google Scholar
Amgarten, D., Braga, L. P. P., da Silva, A. M., e Setubal, J. C. (2018). MARVEL, uno strumento per la predizione di sequenze di batteriofagi in bidoni metagenomici. Front. Genet. 9:304. doi: 10.3389/fgene.2018.00304
PubMed Abstract | CrossRef Full Text | Google Scholar
Kilcher, S., Studer, P., Muessner, C., Klumpp, J., and Loessner, M. J. (2018). Cross-genus reboot di genomi di batteriofagi personalizzati e sintetici in batteri L-form. Proc. Natl. Acad. Sci. U.S.A. 115, 567-572. doi: 10.1073/pnas.1714658115
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemire, S., Yehl, K. M., e Lu, T. K. (2018). Applicazioni basate su fagioli in biologia sintetica. Annu. Rev. Virol. 5, 453-476. doi: 10.1146/annurev-virology-092917-043544
PubMed Abstract | CrossRef Full Text | Google Scholar
Luria, S. E., e Delbrück, M. (1943). Mutazioni di batteri dalla sensibilità al virus alla resistenza al virus. Genetics 28, 491-511.
Google Scholar
Martorell-Marugán, J., Tabik, S., Benhammou, Y., del Val, C., Zwir, I., Herrera, F., et al. (2019). “Deep learning in omics data analysis and precision medicine”, in Computational Biology, ed. H. Husi (Brisbane: Codon Publications), 37-53. doi: 10.15586/computationalbiology.2019.ch3
PubMed Abstract | CrossRef Full Text | Google Scholar
Merabishvili, M., De Vos, D., Verbeken, G., Kropinski, A. M., Vandenheuvel, D., Lavigne, R., et al. (2012). Selezione e caratterizzazione di un candidato batteriofago terapeutico che liscia il ceppo Escherichia coli O104:H4 dall’epidemia 2011 in Germania. PLoS One 7:e52709. doi: 10.1371/journal.pone.0052709
PubMed Abstract | CrossRef Full Text | Google Scholar
Miêdzybrodzki, R., Borysowski, J., Weber-Da̧browska, B., Fortuna, W., Letkiewicz, S., Szufnarowski, K., et al. (2012). Aspetti clinici della terapia fagica. Adv. Virus Res. 83, 73-121. doi: 10.1016/B978-0-12-394438-2.00003-7
PubMed Abstract | CrossRef Full Text | Google Scholar
Palluk, S., Arlow, D. H., de Rond, T., Barthel, S., Kang, J. S., Bector, R., et al. (2018). Sintesi di DNA de novo utilizzando polimerasi-nucleotide coniugati. Nat. Biotechnol. 36, 645-650. doi: 10.1038/nbt.4173
PubMed Abstract | CrossRef Full Text | Google Scholar
Pires, D. P., Cleto, S., Sillankorva, S., Azeredo, J., and Lu, T. K. (2016). Fagi geneticamente ingegnerizzati: una revisione dei progressi negli ultimi dieci anni. Microbiol. Mol. Biol. Rev. 80, 523-543. doi: 10.1128/MMBR.00069-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., De Vos, D., Verbeken, G., Merabishvili, M., Chanishvili, N., Vaneechoutte, M., et al. (2011). Il paradigma della terapia fagica: prêt-à-porter o sur-mesure? Pharm. Res. 28, 934-937. doi: 10.1007/s11095-010-0313-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., Verbeken, G., Ceyssens, P. J., Huys, I., De Vos, D., Ameloot, C., et al. (2018). Il fago magistrale. Viruses 10:E64. doi: 10.3390/v10020064
PubMed Abstract | CrossRef Full Text | Google Scholar
Reyes, A., Haynes, M., Hanson, N., Angly, F. E., Heath, A. C., Rohwer, F., et al. (2010). Virus nel microbiota fecale di gemelli monozigoti e le loro madri. Nature 466, 334-338. doi: 10.1038/nature09199
PubMed Abstract | CrossRef Full Text | Google Scholar
Rustad, M., Eastlund, A., Jardine, P., e Noireaux, V. (2018). Cell-free TXTL sintesi di batteriofago infettivo T4 in una singola reazione in provetta. Synth. Biol. 3:ysy002.
Google Scholar
Schooley, R. T., Biswas, B., Gill, J. J., Hernandez-Morales, A., Lancaster, J., Lessor, L., et al. (2017). Sviluppo e utilizzo di cocktail terapeutici personalizzati basati su batteriofagi per trattare un paziente con un’infezione da Acinetobacter baumannii resistente disseminato. Antimicrob. Agents Chemother. 61:e0954-17. doi: 10.1128/AAC.02221-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Sulakvelidze, A., Alavidze, Z., and Morris, J. G. Jr. (2001). Terapia con batteriofagi. Antimicrob. Agents Chemother. 45, 649-659.
Google Scholar
Suttle, C. A. (2013). Virus: sbloccare la più grande biodiversità sulla Terra. Genome 56, 542-544. doi: 10.1139/gen-2013-0152
PubMed Abstract | CrossRef Full Text | Google Scholar
Taylor, P. W., and Sommer, A. P. (2005). Verso un trattamento razionale delle infezioni batteriche durante i viaggi spaziali estesi. Int. J. Antimicrob. Agents 26, 183-187. doi: 10.1016/j.ijantimicag.2005.06.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiebes, S., Schlesner, M., Brors, B., and Sunyaev, A. (2020). Tecnologia ledger distribuita nella genomica: un appello per l’Europa. Eur. J. Hum. Genet. 28, 139-140. doi: 10.1038/s41431-019-0512-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiel, K. (2004). Vecchio dogma, nuovi trucchi – terapia fagica del 21° secolo. Nat. Biotechnol. 2, 31-36. doi: 10.1038/nbt0104-31
PubMed Abstract | CrossRef Full Text | Google Scholar
Vandersteegen, K., Mattheus, W., Ceyssens, P. J., Bilocq, F., De Vos, D., Pirnay, J.-P., et al. Valutazione microbiologica e molecolare del batteriofago ISP per il controllo dello Staphylococcus aureus. PLoS One 6:e24418. doi: 10.1371/journal.pone.0024418
PubMed Abstract | CrossRef Full Text | Google Scholar
Verbeken, G., Pirnay, J. P., De Vos, D., Jennes, S., Zizi, M., Lavigne, R., et al. (2012). Ottimizzare il quadro normativo europeo per una terapia sostenibile con batteriofagi nella medicina umana. Arch. Immunol. Ther. Exp. 60, 161-172. doi: 10.1007/s00005-012-0175-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Warwick, R. M., Chapman, J., Pruett, T. L., and Wang, H. (2013). Sistemi di codifica coerenti a livello globale per i prodotti medici di origine umana. Bull. World Health Organ. 91, 314-314A. doi: 10.2471/BLT.12.116988
PubMed Abstract | CrossRef Full Text | Google Scholar
Yosef, I., Goren, M. G., Globus, R., Molshanski-Mor, S., e Qimron, U. (2017). Estensione della gamma ospite di particelle di batteriofago per la trasduzione del DNA. Mol. Cell. 66, 721.e3-728.e3. doi: 10.1016/j.molcel.2017.04.025
PubMed Abstract | CrossRef Full Text | Google Scholar