En ontologi är en formell representation av en kunskapsmassa inom en viss domän. Ontologier består vanligtvis av en uppsättning klasser (eller termer eller begrepp) med relationer som fungerar mellan dem. Genontologin (GO) beskriver vår kunskap om den biologiska domänen med avseende på tre aspekter:
| Molekylär funktion | Aktiviteter på molekylär nivå som utförs av genprodukter. Termer för molekylär funktion beskriver aktiviteter som sker på molekylär nivå, t.ex. “katalys” eller “transport”. GO:s molekylära funktionstermer representerar aktiviteter snarare än de enheter (molekyler eller komplex) som utför handlingarna, och anger inte var, när eller i vilket sammanhang handlingen äger rum. Molekylära funktioner motsvarar i allmänhet aktiviteter som kan utföras av enskilda genprodukter (dvs. ett protein eller RNA), men vissa aktiviteter utförs av molekylära komplex som består av flera genprodukter. Exempel på breda funktionella termer är katalytisk aktivitet och transportöraktivitet; exempel på snävare funktionella termer är adenylatcyklasaktivitet eller bindning till Toll-liknande receptorer. För att undvika förvirring mellan genproduktnamn och deras molekylära funktioner, bifogas GO-molekylära funktioner ofta ordet “aktivitet” (ett proteinkinas skulle ha GO-molekylfunktionen proteinkinasaktivitet). | |
| Cellulär komponent | Placeringarna i förhållande till de cellstrukturer i vilka en genprodukt utför en funktion, antingen cellulära kompartment (t.ex, mitokondrion), eller stabila makromolekylära komplex som de är delar av (t.ex. ribosomen). Till skillnad från de andra aspekterna av GO hänvisar cellkomponentklasser inte till processer utan snarare till en cellulär anatomi. | |
| Biologisk process | De större processerna, eller “biologiska program” som åstadkoms av flera molekylära aktiviteter. Exempel på breda termer för biologiska processer är DNA-reparation eller signaltransduktion. Exempel på mer specifika termer är pyrimidinnukleobasbiosyntetisk process eller glukostransmembrantransport. Observera att en biologisk process inte är likvärdig med en väg. För närvarande försöker GO inte representera den dynamik eller de beroenden som skulle krävas för att fullt ut beskriva en väg. |
I ett exempel på GO-annotation kan genprodukten “cytokrom c” beskrivas med den molekylära funktionen oxidoreduktasaktivitet, den biologiska processen oxidativ fosforylering och den cellulära komponenten mitokondriell matris.
GO-vokabulären är utformad för att vara artoberoende och innehåller termer som kan tillämpas på prokaryoter och eukaryoter samt på encelliga och flercelliga organismer.
GO-klasser (även kallade termer)
GO-klasser består av en definition, en etikett, en unik identifierare och flera andra element. Elementen i GO-termer beskrivs här.
GO-grafen
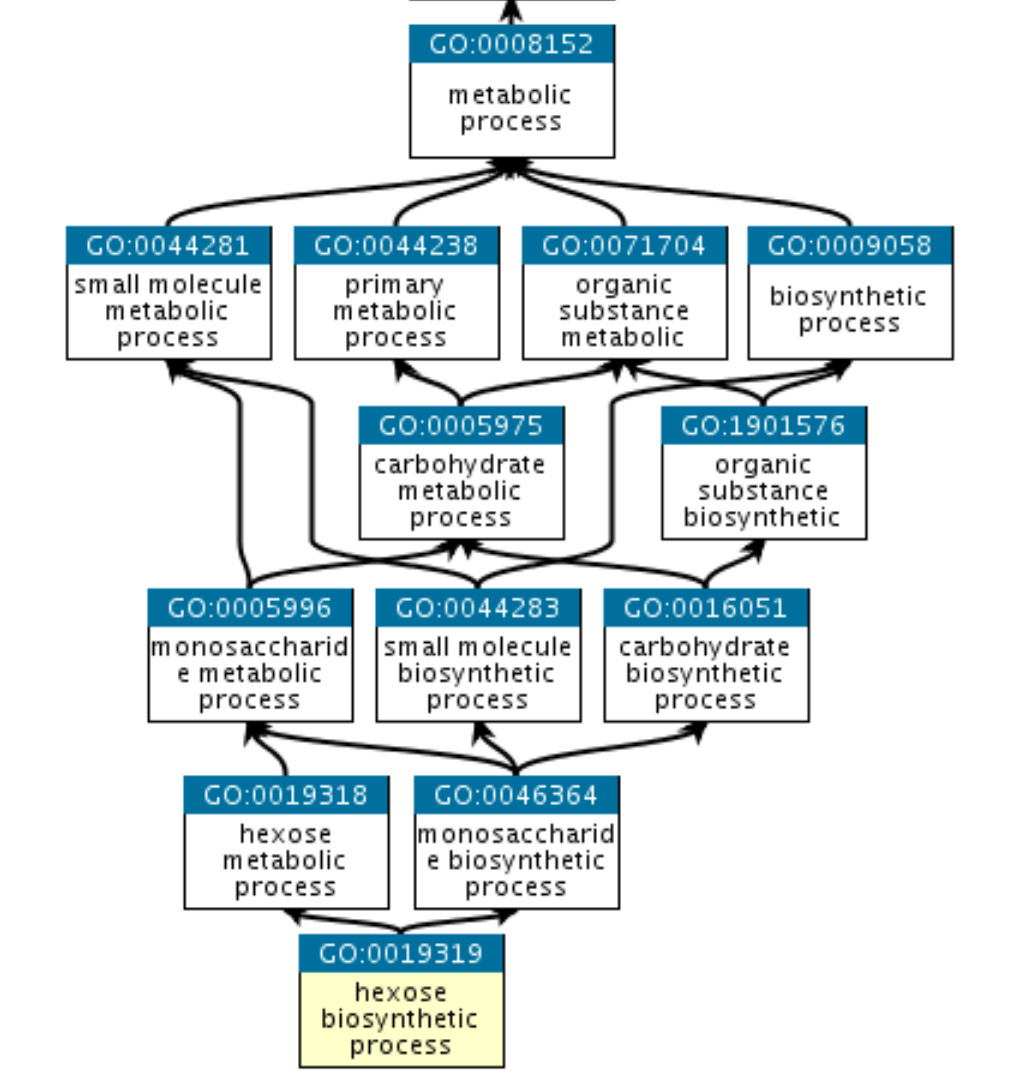
Go:s struktur kan beskrivas i form av en graf, där varje GO-term är en nod och relationerna mellan termerna är kanter mellan noderna. GO är löst hierarkisk, där “barntermer” är mer specialiserade än sina “föräldertermer”, men till skillnad från en strikt hierarki kan en term ha mer än en förälderterm (observera att förälder/barn-modellen inte gäller för alla typer av relationer, se dokumentationen om relationer). Den biologiska processtermen hexose biosynthetic process har till exempel två föräldrar, hexose metabolic process och monosaccharide biosynthetic process. Detta återspeglar det faktum att biosyntetisk process är en undertyp av metabolisk process och hexos är en undertyp av monosackarid.
En ontologi… eller tre?
Som diagrammet ovan antyder representeras de tre GO-domänerna (cellulär komponent, biologisk process och molekylär funktion) var och en av dem av en separat rotontologiterm. Alla termer inom en domän kan spåra sin härstamning till en rotterm, även om det kan finnas många olika vägar via ett varierande antal mellanliggande termer till en ontologirot. De tre rotnoderna är inte besläktade och har ingen gemensam föräldranod, och därför är GO tre ontologier. I dessa fall kan en “falsk” term läggas till som förälder till de tre befintliga rotnoderna.
De tre GO-ontologierna är disjunkta, vilket innebär att det inte finns några relationer mellan termer från de olika ontologierna. Andra relationer, t.ex. part of och regulates, fungerar dock mellan GO-ontologierna. Den molekylära funktionstermen “cyclin-dependent protein kinase activity” är till exempel en del av den biologiska processen “cellcykel”. Mer information om relationer finns här.
GO som dynamisk ontologi
GO syftar till att representera det aktuella kunskapsläget inom biologin, och därför revideras och utökas den ständigt i takt med att den biologiska kunskapen ökar. Ändringar görs varje vecka (de flesta relativt små). Revideringar av ontologin hanteras av ett team av ontologiredaktörer med omfattande erfarenhet av både biologi och datoriserad kunskapsrepresentation. Dessa uppdateringar görs i samarbete mellan GOC:s ontologigrupp och de forskare som begär uppdateringarna. De flesta förfrågningar kommer från forskare som gör GO-annotationer (dessa påverkar vanligtvis endast ett fåtal termer vardera) och från domänexperter inom särskilda biologiska områden (dessa reviderar vanligtvis en hel “gren” av ontologin som omfattar många termer och relationer). Vi inbjuder forskare och datavetare att lämna in önskemål om antingen nya termer, nya relationer eller andra förbättringar av ontologin.
Detaljer om ontologin
- GO-termelement: Beskrivning av GO-termernas format.
- Ontologirelationer: Dokumentation om de relationer mellan termer som används i GO.
- GO-statistik: Statistik för den aktuella versionen och över tid.