ChIP-seq es una técnica maravillosa que nos permite interrogar las interacciones físicas de unión entre la proteína y el ADN utilizando la secuenciación de próxima generación. En este artículo, haré una breve revisión de la ChIP y presentaré la técnica de secuenciación por inmunoprecipitación de la cromatina (ChIP-seq), que combina la ChIP con la secuenciación de nueva generación.
¿Qué es la inmunoprecipitación de la cromatina?
La inmunoprecipitación de la cromatina (ChIP) nos permite determinar los sitios de unión de las proteínas al ADN. La cromatina es el complejo de ADN empaquetado con proteínas histónicas en nucleosomas. La ChIP utiliza los enlaces cruzados reversibles que se producen entre el ADN y las proteínas asociadas mediante la fijación de células o tejidos con formaldehído. La cromatina fijada se cizalla físicamente y se inmunoprecipitan y analizan selectivamente fragmentos de ADN asociados a una proteína concreta. El análisis puede realizarse locus por locus mediante PCR, pero lo más habitual es que el ChIP se analice con microarrays (ChIP-chip) o con secuenciación de nueva generación (ChIP-seq).
¿Cómo funciona el ChIP-chip?
El ChIP-on-chip, o ChIP-chip, combina la inmunoprecipitación de la cromatina con el análisis de microarrays (chip). En 2001, Jason Lieb, del laboratorio de Pat Brown en Stanford, publicó el primer trabajo de ChIP-chip (Lieb 2001). En este método, los fragmentos de ADN que precipitan con una proteína concreta se aplican a un chip de microarrays para su análisis. Esto genera una imagen global de dónde se une la proteína, en contraste con la simple interrogación de sitios de unión individuales por PCR. Aunque se trata de un enfoque revolucionario, la técnica está limitada por las tecnologías de matrices utilizadas. En primer lugar, se necesita una micromatriz para el genoma que se quiere estudiar, y a principios y mediados de la década de 2000 a menudo no era así. En segundo lugar, esos microarrays están limitados por la calidad del genoma de referencia y la capacidad de diseñar sondas que funcionen en un array. En tercer lugar, los arrays están limitados en cuanto a la parte del genoma que pueden representar, y normalmente se reparten por todo el genoma con una resolución de 100 o 1000 pb. También hay problemas sobre el sesgo en la amplificación de los fragmentos de ADN de ChIP, la normalización de los datos de las matrices y la comparabilidad de las plataformas de matrices.
¿Cómo funciona la ChIP-seq?
La secuenciación por inmunoprecipitación de cromatina, o ChIP-seq, combina la ChIP con la secuenciación de próxima generación (Barski 2007, Johnson 2007). Los protocolos de ChIP-seq se han adaptado a partir de los métodos de ChIP-chip: las proteínas se reticulan a su ADN unido mediante un tratamiento con formaldehído, las células se homogeneizan y la cromatina se cizalla y se inmunoprecipita con perlas magnéticas unidas a anticuerpos. El ADN inmunoprecipitado se utiliza entonces como entrada para un protocolo de preparación de bibliotecas de secuenciación de próxima generación, donde se secuencian y analizan los sitios de unión al ADN. Véase en la figura siguiente un resumen del flujo de trabajo de ChIP-seq y un ejemplo de los resultados de ChIP-seq (reproducido con la amable autorización de Dominic Schmidt (Schmidt 2009)).
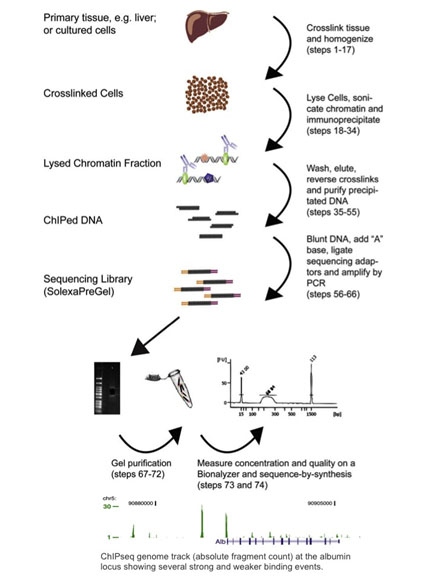
Aunque la mayoría de los aproximadamente 400 artículos publicados hasta ahora se han analizado en la plataforma Illumina, ChIP-seq puede realizarse en cualquier secuenciador de nueva generación (Wold 2008). La ChIP-seq ha sido ampliamente adoptada desde que se informó por primera vez en 2007. De hecho, ha suplantado casi totalmente a ChIP-Chip, ya que permite el análisis de todo el genoma y no tiene las limitaciones discutidas anteriormente.
¿Qué se puede hacer realmente con ChIP-seq
ChIP-seq es una herramienta poderosa y versátil, y hay muchos grandes ejemplos de uso de ChIP-seq en la literatura. He escogido un par de mis favoritos (del trabajo realizado en el centro que dirijo) para ilustrar lo que es posible, y he incluido ejemplos en los que ChIP-seq ha inspirado el desarrollo de nuevos métodos:
- Antoni Hurtado, et al. realizó un knock-down del “factor pionero” FoxA1, lo que resultó en una reducción de la unión del receptor de estrógeno (ER) en más del 50% de los sitios de unión ER conocidos. Demostraron que FoxA1 es un importante regulador de la transcripción mediada por el RE, lo que sugiere que puede ser una nueva e importante diana terapéutica en el cáncer de mama (Hurtado 2011).
- Dominic Shmidt, et al. utilizaron ChIP-seq para investigar la evolución de la unión de factores de transcripción. Se centraron en la unión de CEBPA y HNF4 en el tejido hepático de cinco especies de vertebrados: humano, ratón, perro, zarigüeya y pollo. ChIP-chip habría sido casi imposible dadas las diferentes especies implicadas y las complejidades en el diseño de las sondas (Schmidt 2010).
Las modificaciones posteriores al método ChIP-seq han dado lugar a varios métodos nuevos, incluyendo ChIP-seq para el análisis de la interacción ARN-proteína, así como DNasa-seq y FAIRE-seq, ambos utilizados para identificar regiones reguladoras en el ADN.
En resumen, ChIP-seq es una técnica madura que es preferible a ChIP-chip y puede ser utilizada por casi cualquier grupo con interés en el análisis de la interacción ADN:proteína. ¿Cómo podría utilizar ChIP-seq en su investigación?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of estrogen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specific binding of Rap1 revealed by genome-wide maps of protein-DNA association. Nat Genet. 2001.
Morozova &Marra, Applications of next-generation sequencing technologies in functional genomics. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
¿Te ha ayudado esto? Entonces, por favor, comparte con tu red.