Préface
Ce document offre une vision personnelle de ce qui pourrait être nécessaire pour que la phagothérapie perce enfin comme un outil antibactérien courant. Il est influencé par les échecs et les incertitudes historiques et récents dans le domaine de la phagothérapie et vise à trouver des solutions basées sur les technologies futures et émergentes qui sont censées modeler la science et la société de demain.
Phagothérapie
Les bactériophages (phages) sont les virus des bactéries. Depuis des temps immémoriaux, ils contrôlent la croissance et la propagation de leurs hôtes bactériens. Les virus bactériens sont les entités vivantes les plus omniprésentes dans notre biosphère. On estime qu’il y a 10 millions de fois plus de virus dans les océans qu’il n’y a d’étoiles dans l’univers et si tous les phages de la Terre étaient empilés les uns sur les autres, cette tour s’étendrait plus loin que les 60 galaxies les plus proches (Suttle, 2013). On peut facilement les trouver partout où les bactéries prospèrent : dans les égouts, les rivières ou l’urine et les selles des patients. Les phages des bactéries pathogènes humaines sont le plus souvent composés d’une tête icosaédrique, une sphère à 20 faces plates faite de protéines et contenant un génome d’acide nucléique, à laquelle est attachée une queue protéique. Lorsqu’un phage strictement lytique adhère avec les fibres et les pointes de sa queue à la surface de sa bactérie cible, la gaine de la queue en forme de seringue se contracte et le noyau de la queue est entraîné à travers la paroi cellulaire bactérienne, injectant le génome du phage dans le périplasme de la cellule bactérienne. Immédiatement, l’ADN bactérien et les mécanismes de synthèse des protéines sont détournés pour construire des copies du phage. Certains phages coupent l’ADN bactérien en morceaux. Après une période de latence de quelques minutes à quelques heures, les phages nouvellement formés éclatent de leurs hôtes bactériens, qui sont tués au cours du processus. Les descendants des phages, qui peuvent se compter par centaines par bactérie, partent ensuite à la recherche de nouvelles bactéries hôtes à infecter. En tant que tels, les phages peuvent être considérés comme des antimicrobiens auto-réplicateurs. Il est important de noter que les phages ont évolué de manière à n’infecter que certaines bactéries cibles et qu’ils sont inoffensifs pour les cellules de mammifères.
Les premières preuves de l’existence d’agents de type viral ayant une activité antibactérienne ont été rapportées par le bactériologiste anglais Frederick Twort, et par le microbiologiste franco-canadien Felix d’Hérelle en 1915 et 1917, respectivement (Sulakvelidze et al., 2001). En 1919, d’Hérelle a exploité pour la première fois le potentiel thérapeutique des phages lorsqu’il les a utilisés pour guérir un garçon souffrant de dysenterie à Paris. La phagothérapie a été immédiatement reconnue comme une approche thérapeutique pour traiter les infections bactériennes et la commercialisation de préparations phagothérapeutiques a été entreprise par plusieurs sociétés, comme L’Oréal en Europe et Eli Lilly Company aux États-Unis (Sulakvelidze et al., 2001). En 1923, le microbiologiste géorgien Giorgi Eliava a fondé l’Institut Eliava à Tbilissi, en Géorgie, consacré à la recherche sur la phagothérapie. C’est le début d’un vaste programme de recherche et de développement sur la phagothérapie dans l’ancienne Union soviétique. Cependant, les premières utilisations de la phagothérapie étaient souvent peu fiables et la recherche sur les antibiotiques était également en cours. L’utilisation réussie de la pénicilline pendant la Seconde Guerre mondiale et sa commercialisation mondiale qui s’en est suivie ont conduit les scientifiques occidentaux à se désintéresser de la phagothérapie. Les chercheurs soviétiques, en revanche, ont continué à développer la phagothérapie et à publier leurs résultats, mais en raison du rideau de fer, leurs connaissances et leur expérience ne se sont pas répandues dans le monde (Sulakvelidze et al., 2001). À l’aube du troisième millénaire, le fardeau sanitaire croissant des infections par des bactéries résistantes aux antibiotiques (Cassini et al., 2019) a suscité un regain d’intérêt mondial pour la phagothérapie en tant qu’outil supplémentaire viable pour la gestion clinique des infections bactériennes (Thiel, 2004). Partout dans le monde, des centres de thérapie par les phages sont mis en place, suivant les traces de l’Institut Eliava et de l’unité de thérapie par les phages de l’Institut Hirszfeld à Wrocław, en Pologne (Miêdzybrodzki et al, 2012).
L’année 2035
Fast forward to future Earth of 2035, un monde lugubre caractérisé par la surpopulation humaine, des perturbations majeures de l’écosystème, le réchauffement climatique et la xénophobie.
Alors qu’il trempait dans son bain, le Dr John Iverian, un microbiologiste à la retraite, a soudainement ressenti une piqûre extrêmement douloureuse à l’arrière de son cou, suivie d’un bruit semblable à celui de l’hélice d’un petit avion. Il a passé l’environnement au crible et, du coin de l’œil, il a vu un gros insecte bizarre, doté de longues pattes et antennes effrayantes, assis sur le mur à côté de sa baignoire design. Osuri, le système de gestion domestique du loft d’Iverian situé dans le centre d’Anvers, a identifié l’insecte comme étant la punaise brune marmoréenne Halyomorpha halys, qui s’était répandue dans le monde entier. Le rapport d’Osuri, projeté sur l’un des écrans vidéo de la salle de bains, mentionnait que les personnes mordues présentaient d’abord une petite plaie rouge dans la zone de la piqûre. Lorsqu’elle n’est pas surveillée, la plaie de la morsure gonfle et produit du pus. Fatigué et confus, l’Iverian nonchalant sort de son bain et va se coucher. Il avait décidé de ne pas effectuer la procédure élaborée de traitement de la plaie, qui avait été fortement conseillée par Osuri. Tôt le lendemain matin, cependant, la morsure s’était transformée en une plaie nécrotique présentant des signes évidents d’infection.
Anxieusement, Iverian activa son dispositif Phage-BEAM. BEAM signifiait “Bedside Energized Anti-Microbial”. L’appareil avait la taille et la forme d’une boîte à chaussures, mais avec une apparence plus élégante et polie. Le nom de l’appareil et de son fabricant était inscrit en lettres colorées sur le côté de la boîte émaillée blanche sans soudure. Iverian a sorti un écouvillon de son emballage stérile et l’a passé délicatement sur toute la surface de la plaie, en s’assurant que l’exsudat de la plaie mouille complètement l’embout en ouate de l’écouvillon. Lorsque l’écouvillon s’est approché de la zone “insérer l’échantillon” de la boîte, une petite porte s’est ouverte comme par magie, libérant un hologramme de 10 pouces de haut d’une technicienne de laboratoire, nommée Marcia. Elle a montré à Iverian où introduire l’échantillon. Marcia a été conçue pour guider les clients dans la procédure de test. “Pour de meilleurs résultats, veuillez insérer une nouvelle cartouche de bio-encre phagique, Dr. Iverian,” dit Marcia. Comme c’était le cas pour les imprimantes 2D d’antan, le coût des cartouches de bio-encre était presque aussi élevé que celui de l’appareil Phage-BEAM lui-même. Selon “Business Insider”, la bio-encre de phage était le deuxième liquide le plus cher sur Terre, derrière le Chanel n° 8. Heureusement, en tant qu’un des inventeurs, Iverian avait obtenu le droit de toujours avoir à sa disposition la version la plus récente de cet appareil, y compris un approvisionnement continu en réactifs, et ce gratuitement.
Iverian savait parfaitement comment l’appareil fonctionnait, il n’avait donc pas besoin de l’aide de Marcia. D’abord, l’ADN était extrait de l’embout de l’écouvillon et le métagénome – tout le matériel génétique présent dans l’échantillon, y compris les bactéries infectantes – était déterminé. Ensuite, ces données génétiques ont été envoyées à un serveur sécurisé “Phage XChange” où un algorithme complexe piloté par l’IA a prédit la séquence génomique du phage le plus susceptible de lyser la bactérie infectante identifiée dans le métagénome et censé susciter la réaction immunitaire la plus faible chez le patient. Les données du génome du phage ont été envoyées au dispositif Phage-BEAM, qui a d’abord synthétisé le génome du phage, puis le phage, en utilisant un système propriétaire de production de phages sans bactérie.
Dans l’heure qui suit le prélèvement, le dispositif produirait un produit phagique thérapeutique prêt à l’emploi. Les résultats de la procédure étape par étape seraient transmis sur l’énorme écran vidéo domestique du salon d’Iverian. Assis dans son fauteuil LC2, écoutant la Grande Messe en do mineur de Mozart, Iverian attendait anxieusement que les résultats lui parviennent. Il avait un mauvais pressentiment. Le résultat a donné des frissons à Iverian. Pathogène bactérien identifié : Streptococcus pyogenes souche FE-2033 ! Osuri a immédiatement activé le protocole d’alerte aux infections, envoyant un message au Centre Mondial de Contrôle des Maladies et projetant des informations de base inquiétantes sur la souche bactérienne mortelle mangeuse de chair, considérée comme une menace imminente pour la santé publique depuis 2033. Pendant un moment, Iverian a envisagé d’exciser la plaie infectée et certains tissus sains environnants avec un couteau de cuisine, mais il s’est calmé et a décidé d’attendre et d’appliquer le produit Phage-BEAM imminent. Une heure plus tard, le dispositif Phage-BEAM avait produit des phages synthétiques. Ces phages ont ensuite été mélangés avec les bactéries isolées, dans un module de validation, pour tester leur efficacité in vitro. Quinze minutes plus tard, le feu vert a été donné à Iverian pour commencer le traitement. Iverian a appliqué les phages dans un pansement à base d’hydrogel à libération lente, qui avait d’abord été mélangé avec la suspension concentrée de phages produite par le dispositif Phage-BEAM, et qui contenait également des antibiotiques synergiques. L’hydrogel a temporairement soulagé la douleur, ce qui l’a encore plus calmé. Iverian a répété l’application de l’hydrogel chargé de phages et d’antibiotiques une fois par jour. L’infection de la plaie s’est améliorée dans les 24 heures et après 7 jours, la plaie était presque complètement guérie. L’infection potentiellement mortelle d’Iverian a été traitée avec succès, rapidement et sans quitter son domicile. Mais, au cours des décennies précédentes, il n’était pas certain que la phagothérapie percerait pour devenir un outil antibactérien largement appliqué et cliniquement utile. Le monde médical avait mis du temps à réaliser que la phagothérapie ne devait pas être identique à l’antibiothérapie, et ce principalement en raison des particularités des agents actifs, les phages.
Quelques particularités pertinentes des phages
Les phages présentent un certain nombre de propriétés qui diffèrent des antibiotiques et ont entravé leur développement en tant que produits pharmaceutiques et leur application en thérapie. Premièrement, ils ont tendance à être spécifiques quant aux bactéries qu’ils infectent. Au mieux, ils ciblent une partie considérable d’une seule espèce bactérienne, mais au pire, ils n’infectent qu’un petit nombre de souches au sein d’une même espèce. Les phages thérapeutiques peuvent donc être sélectionnés pour tuer principalement une espèce bactérienne, ou un sous-groupe cliniquement pertinent de celle-ci, et épargner les bactéries bénéfiques du patient (par exemple, la flore intestinale, cutanée ou orale commensale). En revanche, la plupart des antibiotiques utilisés couramment ont un large spectre d’activité, ce qui peut causer des “dommages collatéraux” aux microbiomes commensaux du patient, qui peuvent à leur tour entraîner des effets indésirables tels que la sélection d’espèces bactériennes résistantes aux antibiotiques (par exemple, Clostridium difficile) ou des diarrhées associées aux antibiotiques (Jernberg et al., 2010). L’inconvénient de la spécificité des phages est que les bactéries infectantes doivent être identifiées avant de commencer la phagothérapie. En revanche, dans l’antibiothérapie empirique, on utilise généralement des cocktails d’antibiotiques à large spectre qui agissent sur une multitude de bactéries à Gram positif et à Gram négatif, ainsi que sur divers champignons. Lorsque plus d’informations sont connues (par exemple, à partir d’une culture bactérienne), le traitement peut consister en des antibiotiques à spectre étroit, qui ciblent plus spécifiquement les bactéries ou les champignons identifiés comme étant à l’origine de la maladie.
Deuxièmement, les bactéries et les phages sont impliqués dans une relation hôte-parasite. Les phages strictement lytiques sont omniprésents dans l’environnement et nécessitent la mort de leur hôte bactérien pour compléter leur cycle de vie. Sans hôte, les phages ne peuvent exister. Les phages imposent une sélection pour les hôtes résistants, qui à leur tour imposent une sélection pour les phages efficaces. Il en résulte ce que l’on appelle la “coévolution antagoniste”, une course aux armements entre les bactéries et les phages, caractérisée par l’évolution réciproque de la résistance bactérienne et de l’infectivité des phages (Buckling et Rainey, 2002). Comme avec la plupart des antimicrobiens, les bactéries deviendront donc également résistantes aux phages (Luria et Delbrück, 1943 ; Schooley et al., 2017), mais, contrairement aux antibiotiques statiques, les phages ont la capacité de surmonter la résistance bactérienne (Buckling et Rainey, 2002). Il existe néanmoins des indications selon lesquelles les bactéries et les phages n’augmenteront pas indéfiniment leur résistance et leur infectivité respectives (Fortuna et al., 2019).
Approches de phagothérapie
Au moment du renouveau de la phagothérapie au début des années 2000, deux approches distinctes de phagothérapie avaient été développées (Pirnay et al., 2011). Dans ce que l’on pourrait appeler l’approche à taille unique, des cocktails de phages à large spectre définis, censés cibler la majorité des bactéries suspectées de causer certaines maladies infectieuses, ont été appliqués. Ces cocktails prédéfinis de phages à large spectre ont été développés, produits et testés dans le cadre des modèles pharmaco-économiques actuels, qui ont été conçus pour des médicaments “statiques” tels que les antibiotiques. Cependant, les cocktails de phages à large spectre, actifs contre la plupart des bactéries Gram-positives et/ou Gram-négatives couramment rencontrées dans les maladies infectieuses, devaient contenir de grandes quantités de phages et se sont avérés très difficiles à développer. Il était possible de développer des cocktails de phages à spectre plus étroit, actifs contre une ou quelques espèces bactériennes, à utiliser dans certaines indications et à condition de connaître à l’avance les espèces bactériennes infectantes. Pour certaines espèces bactériennes, comme Staphylococcus aureus, des phages présentant une gamme d’hôtes exceptionnellement large avaient été isolés et caractérisés (Vandersteegen et al., 2011). Dans PhagoBurn, un essai contrôlé randomisé, le succès dans l’obtention d’une réduction durable des charges de Pseudomonas aeruginosa dans les plaies de brûlures était lié à la sensibilité initiale au cocktail de phages (Jault et al., 2019). Cependant, il a été démontré qu’un tiers des patients inclus hébergeaient des souches de P. aeruginosa préexistantes et résistantes au cocktail phagique, qui comprenait pas moins de 12 phages lytiques de P. aeruginosa. En outre, les cocktails de phages initialement conçus pour être efficaces devaient être régulièrement mis à jour (par exemple, complétés par de nouveaux phages) en réponse à l’émergence d’une résistance aux phages ou à l’implication de nouvelles souches circulantes cliniquement pertinentes. Enfin, on ne savait pas si le fait de confronter les bactéries à de fortes concentrations de cocktails de phages fixes entraînerait l’émergence, la propagation et la persistance d’une résistance bactérienne aux phages dans les hôpitaux et dans l’environnement, à l’instar de ce qui s’était produit lors de l’utilisation massive d’antibiotiques.
Dans les concepts de phagothérapie personnalisée, un ou plusieurs phages ont été sélectionnés dans une banque de phages, ou dans l’environnement, et éventuellement adaptés (sélection in vitro de mutants de phages présentant une infectivité accrue) pour infecter plus efficacement les bactéries isolées du site d’infection du patient (Friman et al, 2016). Certains centres de phagothérapie ont mis en place et maintenu de grandes banques de phages thérapeutiques, régulièrement mises à jour avec de nouveaux phages, élargissant et adaptant la gamme d’hôtes de la banque aux populations bactériennes en constante évolution. Les approches personnalisées de la phagothérapie étaient potentiellement plus durables, car seule la bactérie infectante était visée, ce qui réduisait la pression de sélection vers la résistance bactérienne aux phages. Cependant, elles étaient également plus élaborées et plus complexes sur le plan logistique que les approches à taille unique, les souches bactériennes et les phages correspondants étant envoyés dans le monde entier (figure 1). De plus, les concepts de médecine de précision n’étaient, en général, pas compatibles avec la plupart des voies de développement et d’homologation de médicaments (drogues aux États-Unis), qui nécessitaient plusieurs années et des millions d’euros (dollars), et ce pour chaque phage de la banque (Verbeken et al., 2012).
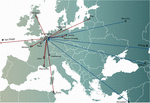
Figure 1. Transferts internationaux de phages depuis (flèches rouges) et vers (flèches bleues) l’Hôpital militaire Reine Astrid (HMRQ) de Bruxelles en vue d’applications cliniques sur la période 2015-2020. Au niveau national, des phages ont été expédiés depuis le QAMH vers cinq hôpitaux universitaires (non illustré). En outre, la sélection des phages correspondants englobait souvent le transfert des isolats bactériens des patients, et cinq patients internationaux (deux de France, deux des Pays-Bas et un de Tunisie) ont été transférés à Bruxelles pour la phagothérapie.
Entrez dans la biologie synthétique
Au début du troisième millénaire, les approches de biologie synthétique avaient été de plus en plus développées pour réduire la spécificité des phages et l’émergence de la résistance bactérienne aux phages (ex, conception guidée par la structure) (Pires et al., 2016 ; Dunne et al., 2019). Par exemple, des plateformes basées sur la levure pour la commutation des protéines des fibres de queue des phages ont été élaborées pour concevoir des phages hybrides avec une gamme d’hôtes plus prévisible et étendue (Ando et al., 2015 ; Yosef et al., 2017) et des stratégies de génie génétique (ex, outils d’édition CRISPR-Cas) ont été développées pour traiter d’autres aspects tels que les interactions négatives patient-phage (par exemple, la réponse immunitaire antiphage) (Brown et al., 2017), l’émergence et la propagation potentielles de mécanismes de résistance aux phages bactériens, et la libération de contenus bactériens nocifs tels que les endotoxines (Hwang et al., 2018). Les génomes de phages synthétiques devaient être réamorcés pour produire des descendants de phages (Barbu et al., 2016 ; Pires et al., 2016), par transformation de formes L d’Escherichia coli ou de Listeria monocytogenes (Kilcher et al., 2018), ou en utilisant des systèmes de transcription-translation sans cellules (TXTL) (Rustad et al., 2018). Les cadres réglementaires occidentaux avaient progressivement commencé à prendre en charge les approches de phagothérapie de précision et personnalisée utilisant des phages d’origine naturelle (Pirnay et al., 2018), des phages fabriqués (Dedrick et al., 2019) et des phages synthétiques.
Le développement de dispositifs de production de phages thérapeutiques ad hoc et sur site, tels que Phage-BEAM, ne s’est pas fait sans heurts, a rappelé Iverian. Pour commencer, il a fallu faire appel à l’intelligence artificielle (IA) pour la mise en correspondance et la conception de phages in silico. L’apprentissage profond (Martorell-Marugán et al., 2019), un sous-ensemble de l’apprentissage automatique, a été choisi pour rechercher des liens entre les génomes bactériens et les génomes des phages infectés, car il était plus facile à adapter à un plus grand nombre d’échantillons. Par exemple, les méthodes d’apprentissage profond ne nécessitaient pas ce que l’on appelle l’extraction de caractéristiques, qui exigerait une annotation au niveau des gènes/protéines des génomes de phages et de bactéries et limiterait les prédictions à certaines relations connues entre les caractéristiques des bactéries et des phages, telles que les structures de fibres de queue des phages se liant à des récepteurs spécifiques de la paroi cellulaire bactérienne. L’inconvénient est qu’elle devait être alimentée par un apport continu de quantités massives de données, reliant les génomes des phages lytiques aux génomes des bactéries hôtes, et c’est là que le bât blesse. Le séquençage du génome entier avait lentement percolé dans la pratique de la microbiologie clinique (American Academy of Microbiology, 2016), mais les instituts de recherche et les entreprises pharmaceutiques n’étaient pas enclins à soumettre leurs données à une base de données centralisée unique, et aucun investisseur n’a été trouvé désireux et capable d’acquérir les données disponibles et/ou de produire des quantités suffisantes de nouvelles données. Un deuxième obstacle à surmonter était l’absence de synthèse rapide, fiable et abordable de grandes molécules d’ADN. Les premières techniques de synthèse d’ADN étaient basées sur la chimie organique et produisaient des molécules d’ADN relativement petites. La synthèse de novo des génomes de phages nécessitait l’assemblage de plusieurs fragments de génome (Barbu et al., 2016 ; Pires et al., 2016 ; Lemire et al., 2018) dans la levure Saccharomyces cerevisiae, en utilisant des chromosomes artificiels de levure (Ando et al., 2015), ou un assemblage chimique (Gibson et al., 2009). Le développement d’une nouvelle technique de synthèse de l’ADN, basée sur les enzymes de synthèse de l’ADN présentes dans les cellules du système immunitaire (Palluk et al., 2018), a facilité la synthèse du génome des phages. Enfin, certains obstacles ont dû être surmontés pour développer des systèmes génériques de production de phages sans cellules, capables de produire des phages à des titres élevés et présentant les mêmes niveaux d’infectivité bactérienne que leurs analogues naturels.
La percée
Le problème majeur était qu’il s’est avéré très difficile de collecter les quantités massives de séquences de phages et de génomes bactériens liés nécessaires aux algorithmes d’IA d’apprentissage profond pour prédire et/ou concevoir des séquences de phages avec un niveau de précision thérapeutiquement acceptable. Iverian se souvient que la véritable percée a eu lieu lorsque l’organisation à but non lucratif “Phage XChange” a lancé sa plateforme mondiale de gouvernance des phages du même nom pour créer une chaîne d’approvisionnement en phages efficace, normalisée, durable et éthique (figure 2). Phage XChange se compose principalement d’un module d’intelligence artificielle et d’un grand livre distribué (DL) (Thiebes et al., 2020). Le module d’IA de la plateforme a analysé les génomes bactériens et phagiques liés afin de prédire et de concevoir des phages puissants pour les clients. Il a également prédit quels agents pathogènes bactériens nécessitaient l’attention la plus urgente, en se basant sur l’Internet des objets (IoT) et le Big Data, ainsi que sur les informations fournies par les organisations internationales de santé publique, telles que l’Organisation mondiale de la santé (OMS) et les centres nationaux de contrôle des maladies. Ces données ont orienté le système vers l’isolement et la caractérisation des phages les plus urgents.
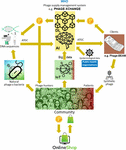
Figure 2. Vision de la façon dont la chaîne d’approvisionnement en phages pourrait être organisée en 2035. IA, intelligence artificielle ; ATGC, séquence d’ADN ; BEAM, bedside energized anti-microbial ; DL, distributed ledger ; IoT, Internet of Things ; P, PhageCoin ; OMS, Organisation mondiale de la santé.
Le module DL de la plateforme a assuré une entrée suffisante, qualitative et enregistrée des séquences génomiques phagiques/bactériennes liées au module IA et ditto la fourniture des séquences phagiques aux clients, conformément aux dispositions du protocole de Nagoya (Expert round table on acceptance and re-implementation of bacteriophage therapy et al, 2018). Le DL a enregistré de manière immuable tous les détails des parties prenantes (par exemple, les fournisseurs de données, les services de séquençage et les clients), des transactions et des contrats. Il a également enregistré la qualité, les spécifications et le poids exacts du matériel fourni. Un algorithme a déterminé la non-redondance et a estimé le poids (par exemple, la virulence et la gamme d’hôtes des phages) et la désirabilité du matériel soumis. Les phages ciblant les pathogènes bactériens émergents étaient bien sûr les plus recherchés. La plupart des problèmes de brevet ont été évités. Le DL a fait office de registre des paiements pour s’assurer que toutes les parties étaient payées à temps et équitablement. Un certain nombre de PhageCoins (la crypto-monnaie de la plateforme) ont été attribués aux fournisseurs en fonction de la qualité, du poids et de l’intérêt du matériel fourni. Les clients qui extrayaient des résultats de prédiction (séquences de phages) par le biais de la DL payaient un montant de PhageCoins, proportionnel à la valeur estimée des phages. Ces PhageCoins ont été utilisés pour maintenir la DL, pour assurer un afflux suffisant et continu de matériel, et pour étendre les données sur la virulence des phages et la gamme d’hôtes (en associant les phages aux bactéries). Une injection supplémentaire de fonds et une incitation à fournir du matériel ont été trouvées chez les producteurs et fournisseurs de toutes sortes de biens. Avec les PhageCoins gagnés instantanément, les fournisseurs de phages pouvaient acheter en ligne toutes sortes de produits à des prix fortement réduits, du matériel de laboratoire et scolaire aux articles de sport. Ces produits étaient fournis grâce au parrainage d’entreprises. Plusieurs entreprises établies ont parrainé PhageXchange en échange de réductions fiscales, de publicité et de l’image d’une marque socialement responsable. Le poids du matériel fourni, et donc sa valeur, étaient initialement sous-évalués, mais étaient réévalués à intervalles réguliers (itération) et les fournisseurs se voyaient attribuer davantage de PhageCoins lorsque cela était justifié. Bien qu’utile dès son introduction, la plateforme n’a connu un réel succès que lorsqu’elle a été placée sous la protection de l’OMS, par analogie avec le système mondial de traçabilité, de transparence, de vigilance et de surveillance des médicaments d’origine humaine (Warwick et al., 2013). Un accord formel entre Phage XChange et l’OMS a renforcé la confiance internationale dans la durabilité à long terme de la plateforme et la protection contre une exploitation commerciale non éthique. La recherche de phages thérapeutiques puissants est rapidement devenue un effort communautaire visant à résoudre la crise de la résistance aux antibiotiques, avec des “chasseurs de phages” indépendants, des écoles, des groupes de scouts, des villages sur les rives de l’Amazone, etc. qui isolent et soumettent des phages à Phage XChange, en échange de PhageCoins. En marge de cela, diverses entreprises et institutions ont développé des équipements et des services périphériques, tels que des kits d’isolement de phages et des plateformes de séquençage et de synthèse de phages (par exemple, le dispositif Phage-BEAM). En prévision de ces dispositifs, des solutions intermédiaires ont été proposées, dans lesquelles les phages eux-mêmes étaient obtenus par le biais de la DL.
Epilog
Cette vision de l’avenir de la phagothérapie offre une issue optimiste à la crise de la résistance aux antibiotiques. La production ad hoc et sur site de phages synthétiques, liée à un système de gestion des phages mondial et communautaire, s’est avérée être une arme supplémentaire bienvenue et abordable (pour les systèmes de sécurité sociale) dans la lutte contre les infections bactériennes résistantes aux antibiotiques. Toutefois, il ne s’agissait pas d’une solution miracle, mais d’un complément synergique aux antimicrobiens établis. La production instantanée et acellulaire de phages synthétiques, qu’ils soient conçus ou non, présentait des avantages considérables par rapport aux phages naturels produits classiquement (dans des hôtes bactériens) :
(i) Il n’était pas nécessaire de maintenir des banques physiques de phages thérapeutiques et d’envoyer les isolats bactériens du patient et les phages thérapeutiques correspondants dans le monde entier.
(ii) Les phages synthétiques contre les bactéries causant des menaces éminentes pour la santé publique, telles que l’épidémie d’E. coli O104:H4 de 2011 en Allemagne (Merabishvili et al., 2012), ou des bactéries (soupçonnées d’être) utilisées pour le bioterrorisme (Joñczyk-Matysiak et al., 2014) pourraient être produites en temps opportun sur place.
(iii) Les phages contre les bactéries causant des maladies potentiellement létales, pour lesquelles aucune souche hôte de production non létale n’était disponible et dont la propagation nécessitait auparavant des précautions de bioconfinement de niveau de biosécurité 3 (bsl-3), pourraient être synthétisés dans des conditions bsl-1.
(iv) Lorsqu’aucun phage n’a pu être isolé à partir des sites d’échantillonnage, par exemple parce que les souches hôtes bactériennes utilisées dans les techniques d’isolement n’étaient pas sensibles aux phages souhaités, les séquences génomiques (prédites) des phages, extraites des données métagénomiques (Reyes et al, 2010 ; Amgarten et al., 2018), ont pu être utilisées pour produire des phages synthétiques.
(v) Les préparations de phages synthétiques ne contenaient aucune (ou de plus petites quantités de) molécules pouvant avoir un impact négatif sur les patients (par exemple, des endotoxines).
(vi) Des dispositifs ont été adaptés pour produire des phages synthétiques pendant les voyages spatiaux prolongés et la colonisation de l’espace (Taylor et Sommer, 2005).
Il y a peu de chances que ces prédictions se réalisent. Il est probablement trop peu clairvoyant de penser qu’un effort communautaire, soutenu par des organisations de santé publique et géré par une plateforme mondiale, durable et éthique, pourrait être au cœur d’une solution à la crise mondiale actuelle de la résistance aux antibiotiques. Certaines parties du système proposé, telles que la production sans cellules de phages synthétiques à l’aide d’un appareil de chevet, ont une chance raisonnable d’être réalisées, tandis que d’autres éléments, tels que le parrainage par des entreprises, resteront probablement limités au domaine de la science-fiction. Vous pouvez dire que je suis un rêveur, alors n’hésitez pas à me réveiller en 2035 pour me confronter à la réalité !
Contributions des auteurs
J-PP a conçu la vision et rédigé le manuscrit.
Financement
Les coûts de publication ont été couverts par la “Société Scientifique du Service Médical Militaire – Wetenschappelijke Vereniging van de Militaire Medische Dienst”.
Conflit d’intérêt
L’auteur déclare que la recherche a été menée en l’absence de toute relation commerciale ou financière qui pourrait être interprétée comme un conflit d’intérêt potentiel.
L’éditeur traitant a déclaré avoir été coauteur dans le passé avec l’auteur.
Remerciements
La vision personnelle, ou le rêve, développé dans ce manuscrit est né des interactions avec de nombreux collègues chercheurs au cours des 15 dernières années. Il est impossible de les nommer tous, mais il ne serait pas juste de prendre tous les crédits seul. J’ai donc décidé d’en citer quelques-uns ici (par ordre alphabétique), au risque – ou mieux, avec la certitude – d’oublier certains influenceurs importants : Joana Azeredo, Nata Bakuradze, Bob Blasdel, Dimitri Boeckaerts, Angus Buckling, Yves Briers, Pieter-Jan Ceyssens, Nina Chanishvili, Laurent Debarbieux, Sarah Djebara, Dorien Dams, Daniel De Vos, Quirin Emslander, Alan Fauconnier, Ville Friman, Andrzej Górski, Téa Glonti, Nino Grdzelishvili, Serge Jennes, Elene Kakabadze, Betty Kutter, Rob Lavigne, Cédric Lood, Alice Maestri, Khatuna Makalatia, Maya Merabishvili, Tobi Nagel, Thomas Rose, Patrick Soentjens, Michiel Stock, Rüdiger Trojok, An Van den Bossche, Mario Vaneechoutte, Gilbert Verbeken et Kilian Vogele.
Académie américaine de microbiologie (2016). Applications du séquençage de nouvelle génération microbien clinique : Rapport sur un colloque de l’Académie américaine de microbiologie tenu à Washington, DC, en avril 2015. Washington, DC : American Society for Microbiology.
Google Scholar
Amgarten, D., Braga, L. P. P., da Silva, A. M., et Setubal, J. C. (2018). MARVEL, un outil de prédiction des séquences de bactériophages dans les bacs métagénomiques. Front. Genet. 9:304. doi : 10.3389/fgene.2018.00304
PubMed Abstract | Ref Full Text | Google Scholar
Kilcher, S., Studer, P., Muessner, C., Klumpp, J., et Loessner, M. J. (2018). Redémarrage transgénique de génomes de bactériophages synthétiques sur mesure dans des bactéries de forme L. Proc. Natl. Acad. Sci. U.S.A. 115, 567-572. doi : 10.1073/pnas.1714658115
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemire, S., Yehl, K. M., et Lu, T. K. (2018). Applications basées sur les phages en biologie synthétique. Annu. Rev. Virol. 5, 453-476. doi : 10.1146/annurev-virology-092917-043544
PubMed Abstract | CrossRef Full Text | Google Scholar
Luria, S. E., et Delbrück, M. (1943). Mutations des bactéries de la sensibilité au virus à la résistance au virus. Genetics 28, 491-511.
Google Scholar
Martorell-Marugán, J., Tabik, S., Benhammou, Y., del Val, C., Zwir, I., Herrera, F., et al. (2019). ” Deep learning in omics data analysis and precision medicine “, dans Computational Biology, éd. H. Husi (Brisbane : Codon Publications), 37-53. doi : 10.15586/computationalbiology.2019..ch3
PubMed Abstract | CrossRef Full Text | Google Scholar
Merabishvili, M., De Vos, D., Verbeken, G., Kropinski, A. M., Vandenheuvel, D., Lavigne, R., et al. (2012). Sélection et caractérisation d’un bactériophage thérapeutique candidat qui lyse la souche Escherichia coli O104:H4 de l’épidémie de 2011 en Allemagne. PLoS One 7:e52709. doi : 10.1371/journal.pone.0052709
PubMed Abstract | CrossRef Full Text | Google Scholar
Miêdzybrodzki, R., Borysowski, J., Weber-Da̧browska, B., Fortuna, W., Letkiewicz, S., Szufnarowski, K., et al. (2012). Aspects cliniques de la phagothérapie. Adv. Virus Res. 83, 73-121. doi : 10.1016/B978-0-12-394438-2.00003-7
PubMed Abstract | CrossRef Full Text | Google Scholar
Palluk, S., Arlow, D. H., de Rond, T., Barthel, S., Kang, J. S., Bector, R., et al. (2018). Synthèse d’ADN de novo à l’aide de conjugués polymérase-nucléotide. Nat. Biotechnol. 36, 645-650. doi : 10.1038/nbt.4173
PubMed Abstract | CrossRef Full Text | Google Scholar
Pires, D. P., Cleto, S., Sillankorva, S., Azeredo, J., et Lu, T. K. (2016). Les phages génétiquement modifiés : une revue des avancées au cours de la dernière décennie. Microbiol. Mol. Biol. Rev. 80, 523-543. doi : 10.1128/MMBR.00069-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., De Vos, D., Verbeken, G., Merabishvili, M., Chanishvili, N., Vaneechoutte, M., et al. (2011). Le paradigme de la phagothérapie : prêt-à-porter ou sur-mesure ? Pharm. Res. 28, 934-937. doi : 10.1007/s11095-010-0313-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., Verbeken, G., Ceyssens, P. J., Huys, I., De Vos, D., Ameloot, C., et al. (2018). Le phage magistral. Virus 10:E64. doi : 10.3390/v10020064
PubMed Abstract | CrossRef Full Text | Google Scholar
Reyes, A., Haynes, M., Hanson, N., Angly, F. E., Heath, A. C., Rohwer, F., et al. (2010). Virus dans le microbiote fécal de jumeaux monozygotes et de leurs mères. Nature 466, 334-338. doi : 10.1038/nature09199
PubMed Abstract | CrossRef Full Text | Google Scholar
Rustad, M., Eastlund, A., Jardine, P., et Noireaux, V. (2018). Synthèse TXTL sans cellules du bactériophage T4 infectieux dans une seule réaction en tube à essai. Synth. Biol. 3:ysy002.
Google Scholar
Schooley, R. T., Biswas, B., Gill, J. J., Hernandez-Morales, A., Lancaster, J., Lessor, L., et al. (2017). Développement et utilisation de cocktails thérapeutiques personnalisés à base de bactériophages pour traiter un patient atteint d’une infection à Acinetobacter baumannii résistante disséminée. Antimicrob. Agents Chemother. 61:e0954-17. doi : 10.1128/AAC.02221-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Sulakvelidze, A., Alavidze, Z., et Morris, J. G. Jr. (2001). La thérapie par bactériophages. Antimicrob. Agents Chemother. 45, 649-659.
Google Scholar
Suttle, C. A. (2013). Virus : déverrouillage de la plus grande biodiversité sur Terre. Genome 56, 542-544. doi : 10.1139/gen-2013-0152
PubMed Abstract | CrossRef Full Text | Google Scholar
Taylor, P. W., et Sommer, A. P. (2005). Vers un traitement rationnel des infections bactériennes pendant les voyages spatiaux prolongés. Int. J. Antimicrob. Agents 26, 183-187. doi : 10.1016/j.ijantimicag.2005.06.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiebes, S., Schlesner, M., Brors, B., et Sunyaev, A. (2020). Technologie de grand livre distribué en génomique : un appel à l’Europe. Eur. J. Hum. Genet. 28, 139-140. doi : 10.1038/s41431-019-0512-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiel, K. (2004). Vieux dogme, nouveaux trucs – la phagothérapie du 21e siècle. Nat. Biotechnol. 2, 31-36. doi : 10.1038/nbt0104-31
PubMed Abstract | CrossRef Full Text | Google Scholar
Vandersteegen, K., Mattheus, W., Ceyssens, P. J., Bilocq, F., De Vos, D., Pirnay, J.-P., et al. (2011). Évaluation microbiologique et moléculaire du bactériophage ISP pour le contrôle de Staphylococcus aureus. PLoS One 6:e24418. doi : 10.1371/journal.pone.0024418
PubMed Abstract | CrossRef Full Text | Google Scholar
Verbeken, G., Pirnay, J. P., De Vos, D., Jennes, S., Zizi, M., Lavigne, R., et al. (2012). Optimiser le cadre réglementaire européen pour une thérapie durable par bactériophages en médecine humaine. Arch. Immunol. Ther. Exp. 60, 161-172. doi : 10.1007/s00005-012-0175-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Warwick, R. M., Chapman, J., Pruett, T. L., et Wang, H. (2013). Systèmes de codage globalement cohérents pour les produits médicaux d’origine humaine. Bull. Organisation mondiale de la santé. 91, 314-314A. doi : 10.2471/BLT.12.116988
PubMed Abstract | CrossRef Full Text | Google Scholar
Yosef, I., Goren, M. G., Globus, R., Molshanski-Mor, S., et Qimron, U. (2017). Extension de la gamme d’hôtes des particules de bactériophages pour la transduction de l’ADN. Mol. Cell. 66, 721.e3-728.e3. doi : 10.1016/j.molcel.2017.04.025
PubMed Abstract | CrossRef Full Text | Google Scholar
.