ChIP-seq is een prachtige techniek waarmee we de fysieke bindingsinteracties tussen eiwitten en DNA kunnen onderzoeken met behulp van next-generation sequencing. In dit artikel geef ik een kort overzicht van ChIP en introduceer ik de chromatine immunoprecipitatie sequencing techniek (ChIP-seq), die ChIP combineert met next-generation sequencing.
Wat is chromatine immunoprecipitatie?
Chromatine immunoprecipitatie (ChIP) stelt ons in staat om eiwit-bindende plaatsen op DNA te bepalen. Chromatine is het complex van DNA dat met histon-eiwitten is verpakt in nucleosomen. ChIP maakt gebruik van omkeerbare cross-links die worden gemaakt tussen DNA en geassocieerde eiwitten door formaldehyde fixatie van cellen of weefsel. Het gefixeerde chromatine wordt fysisch geschoren en DNA-fragmenten die met een bepaald eiwit geassocieerd zijn, worden selectief geïmmunoprecipiteerd en geanalyseerd. Analyse kan plaatsvinden op een locus-per-locus basis met behulp van PCR, maar vaker wordt ChIP ondervraagd met microarrays (ChIP-chip) of next-generation sequencing (ChIP-seq).
Hoe werkt ChIP-chip?
ChIP-on-chip, of ChIP-chip, combineert chromatine-immunoprecipitatie met microarray (chip) analyse. In 2001 publiceerde Jason Lieb in het lab van Pat Brown in Stanford het eerste ChIP-chip artikel (Lieb 2001). Bij deze methode worden de DNA-fragmenten die neerslaan met een bepaald eiwit voor analyse op een microarray-chip aangebracht. Dit geeft een globaal beeld van waar het eiwit bindt, in tegenstelling tot het ondervragen van afzonderlijke bindingsplaatsen met PCR. Hoewel dit een revolutionaire benadering was, wordt de techniek beperkt door de gebruikte array-technologieën. In de eerste plaats heb je een microarray nodig voor het genoom dat je wilt bestuderen, en in het begin en het midden van de jaren 2000 was dat vaak niet het geval. Ten tweede zijn die microarrays beperkt door de kwaliteit van het referentiegenoom en de mogelijkheid om probes te ontwerpen die op een array werken. Ten derde zijn arrays beperkt in de mate waarin het genoom kan worden gerepresenteerd, en meestal worden ze over het genoom verdeeld met een resolutie van 100 of 1000 bp. Er zijn ook problemen met de bias in de amplificatie van de ChIP DNA-fragmenten, normalisatie van array-gegevens, en de vergelijkbaarheid van array-platforms.
Hoe werkt ChIP-seq?
Chromatine immunoprecipitatie sequencing, of ChIP-seq, combineert ChIP met next-generation sequencing (Barski 2007, Johnson 2007). ChIP-seq protocollen zijn aangepast van ChIP-chip methoden: eiwitten worden cross-linked aan hun gebonden DNA door formaldehyde behandeling, cellen worden gehomogeniseerd, en chromatine wordt geschud en immunoprecipitated met antilichaam-gebonden magnetische kralen. Het geïmmunoprecipiteerde DNA wordt vervolgens gebruikt als input voor een next-generation sequencing library prep protocol, waar het wordt gesequeneerd en geanalyseerd op DNA-bindingsplaatsen. Zie onderstaande figuur voor een samenvatting van de ChIP-seq-workflow en een voorbeeld van ChIP-seq-resultaten (overgenomen met toestemming van Dominic Schmidt (Schmidt 2009)).
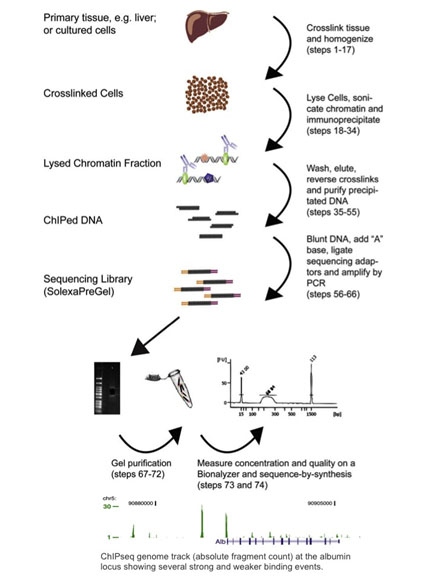
Hoewel de meeste van de ongeveer 400 tot dusver gepubliceerde papers zijn geanalyseerd op het Illumina-platform, kan ChIP-seq worden uitgevoerd op elke next-generation sequencer (Wold 2008). ChIP-seq is op grote schaal toegepast sinds het in 2007 voor het eerst werd gemeld. In feite heeft het bijna volledig ChIP-Chip verdrongen, omdat het genoom-brede analyse mogelijk maakt en niet de beperkingen heeft die hierboven zijn besproken.
Wat kun je eigenlijk doen met ChIP-seq
ChIP-seq is een krachtig instrument en een veelzijdig instrument, en er zijn veel geweldige voorbeelden van het gebruik van ChIP-seq in de literatuur. Ik heb een paar van mijn favorieten uitgekozen (van werk dat is uitgevoerd in de kernfaciliteit die ik beheer) om te illustreren wat er mogelijk is, en voorbeelden opgenomen waar ChIP-seq de ontwikkeling van nieuwe methoden heeft geïnspireerd:
- Antoni Hurtado, et al. voerden knock-down uit van de FoxA1 “pionierfactor”, wat resulteerde in verminderde binding door de oestrogeenreceptor (ER) op meer dan 50% van de bekende ER-bindingsplaatsen. Zij toonden aan dat FoxA1 een belangrijke regulator is van ER-gemedieerde transcriptie, wat suggereert dat het een nieuw en belangrijk therapeutisch doelwit kan zijn bij borstkanker (Hurtado 2011).
- Dominic Shmidt, et al. gebruikten ChIP-seq om de evolutie van binding van transcriptiefactoren te onderzoeken. Zij richtten zich op CEBPA en HNF4 binding in het leverweefsel van vijf gewervelde diersoorten: mens, muis, hond, opossum en kip. ChIP-chip zou bijna onmogelijk zijn geweest gezien de verschillende betrokken soorten en de complexiteit bij het ontwerpen van probes (Schmidt 2010).
Verdere aanpassingen aan de ChIP-seq methode hebben geleid tot verschillende nieuwe methoden, waaronder ChIP-seq voor RNA-eiwit interactie analyse, evenals DNase-seq en FAIRE-seq, die beide worden gebruikt om regulerende regio’s in DNA te identificeren.
Samenvattend, ChIP-seq is een volwassen techniek die de voorkeur verdient boven ChIP-chip en kan worden gebruikt door bijna elke groep met interesse in DNA:Eiwit interactie analyse. Hoe zou u ChIP-seq in uw onderzoek kunnen gebruiken?
Barski et al, High-resolution profiling of histone methylations in the human genome. Cell 129 (2007).
Hurtado et al, FOXA1 is a key determinant of oestrogeen receptor function and endocrine response.Nature Genetics (2011).
Johnson et al, Genome-wide mapping of in vivo protein-DNA interactions. Science (2007).
Lee et al, Chromatin immunoprecipitation and microarray-based analysis of protein location. Nat. Protoc (2006).
Lieb et al, Promoter-specifieke binding van Rap1 onthuld door genoom-brede kaarten van eiwit-DNA associatie. Nat Genet. 2001.
Morozova & Marra, Applications of next-generation sequencing technologies in functional genomics. Genomics (2008).
Schmidt et al, ChIP-seq: Using high-throughput sequencing to discover protein-DNA interactions. Methods (2009).
Schmidt et al, Five-Vertebrate ChIP-seq Reveals the Evolutionary Dynamics of Transcription Factor Binding Science (2010).
Wold & Myers, Sequence census methods for functional genomics. Nat. Methods (2008).
Heeft dit u geholpen? Deel het dan met uw netwerk.