Przedmowa
Ta praca przedstawia osobistą wizję tego, co może być potrzebne, aby terapia fagowa w końcu przebiła się jako główne narzędzie antybakteryjne. Jest ona pod wpływem historycznych i niedawnych niepowodzeń i niepewności w dziedzinie terapii fagowej i ma na celu znalezienie rozwiązań opartych na przyszłych i powstających technologiach, które mają modelować naukę i społeczeństwo jutra.
Terapia fagowa
Bakteriofagi (fagi) są wirusami bakterii. Od niepamiętnych czasów kontrolują one wzrost i rozprzestrzenianie się swoich bakteryjnych gospodarzy. Wirusy bakteryjne są najbardziej wszechobecnymi istotami żywymi w naszej biosferze. Szacuje się, że w oceanach znajduje się 10 milionów razy więcej wirusów niż jest gwiazd we wszechświecie, a gdyby wszystkie fagi na Ziemi zostały ułożone jeden na drugim, ta wieża rozciągałaby się dalej niż najbliższe 60 galaktyk (Suttle, 2013). Można je łatwo znaleźć wszędzie tam, gdzie rozwijają się bakterie: w kanałach ściekowych, rzekach lub w moczu i stolcu pacjentów. Fagi ludzkich patogenów bakteryjnych składają się najczęściej z icosaedrycznej głowy, kuli o 20 płaskich powierzchniach zbudowanej z białek i zawierającej genom kwasu nukleinowego, do której przyczepiony jest białkowy ogon. Kiedy fag ściśle lityczny przylega włóknami ogona i kolcami do powierzchni docelowej bakterii, strzykawkowata otoczka ogona kurczy się, a rdzeń ogona jest wbijany w ścianę komórkową bakterii, wstrzykując genom faga do peryplazmy komórki bakteryjnej. Natychmiast bakteryjna maszyneria syntezy DNA i białek zostaje porwana do budowy kopii faga. Niektóre fagi tną DNA bakterii na kawałki. Po okresie utajenia, trwającym od kilku minut do kilku godzin, nowo powstałe fagi odrywają się od swoich bakteryjnych gospodarzy, którzy giną w tym procesie. Potomstwo fagów, których liczba może sięgać setek na bakterię, wyrusza na poszukiwanie nowych bakterii gospodarzy, aby je zainfekować. W związku z tym fagi mogą być uważane za samoreplikujące się środki przeciwdrobnoustrojowe. Co ważne, fagi ewoluowały tak, aby infekować tylko określone bakterie docelowe i są nieszkodliwe dla komórek ssaków.
Wcześniejsze dowody na istnienie czynników wirusopodobnych o aktywności przeciwbakteryjnej zostały zgłoszone przez angielskiego bakteriologa Fredericka Tworta oraz francusko-kanadyjskiego mikrobiologa Felixa d’Hérelle odpowiednio w 1915 i 1917 roku (Sulakvelidze i in., 2001). W 1919 r. d’Hérelle po raz pierwszy wykorzystał potencjał terapeutyczny fagów, lecząc za ich pomocą chłopca cierpiącego na czerwonkę w Paryżu. Terapia fagowa została natychmiast uznana za metodę leczenia infekcji bakteryjnych, a komercjalizacja preparatów do terapii fagowej została podjęta przez kilka firm, takich jak L’Oréal w Europie i Eli Lilly Company w Stanach Zjednoczonych (Sulakvelidze i in., 2001). W 1923 r. gruziński mikrobiolog Giorgi Eliava założył w Tbilisi (Gruzja) Instytut Eliava, poświęcony badaniom nad terapią fagową. Był to początek szeroko zakrojonych badań i rozwoju terapii fagowej na terenie byłego Związku Radzieckiego. Jednak wczesne zastosowania terapii fagowej były często zawodne, a badania nad antybiotykami również trwały. Udane zastosowanie penicyliny podczas II wojny światowej i jej późniejszy światowy marketing spowodowały, że zachodni naukowcy stracili zainteresowanie terapią fagową. Z kolei badacze radzieccy nadal rozwijali terapię fagową i publikowali swoje wyniki, ale z powodu żelaznej kurtyny ich wiedza i doświadczenie nie rozprzestrzeniały się na cały świat (Sulakvelidze i in., 2001). U progu trzeciego tysiąclecia, rosnące obciążenie zdrowotne związane z zakażeniami bakteriami opornymi na antybiotyki (Cassini i in., 2019) spowodowało ponowne zainteresowanie terapią fagową jako realnym dodatkowym narzędziem w klinicznym leczeniu zakażeń bakteryjnych (Thiel, 2004). Na całym świecie powstają ośrodki terapii fagowej, idąc w ślady Instytutu Eliava i oddziału terapii fagowej w Instytucie Hirszfelda we Wrocławiu (Międzybrodzki i wsp., 2012).
Rok 2035
Przenosimy się w przyszłość do Ziemi roku 2035, ponurego świata charakteryzującego się przeludnieniem, poważnymi zakłóceniami ekosystemu, globalnym ociepleniem i ksenofobią.
Podczas kąpieli w wannie dr John Iverian, emerytowany mikrobiolog, nagle poczuł niezwykle bolesne ukłucie w tylnej części szyi, po którym nastąpił dźwięk przypominający śmigło małego samolotu. Rozejrzał się po otoczeniu i kątem oka zobaczył dziwnego, dużego owada z długimi, pełzającymi odnóżami i antenami siedzącego na ścianie obok jego designerskiej wanny. Osuri, system zarządzania domem w lofcie Iveriana w centrum Antwerpii, zidentyfikował owada jako brązowego marmorowatego robaka cuchnącego Halyomorpha halys, który rozprzestrzenił się na całym świecie. Raport Osuri’ego, wyświetlany na jednym z ekranów wideo w łazience, wspominał, że ludzie, którzy zostali ugryzieni, początkowo odczuwali małe, czerwone rany w miejscu ugryzienia. Pozostawiona bez opieki, rana po ugryzieniu puchła i wytwarzała ropę. Zmęczony i zamroczony, niechętny Iverian wyszedł z kąpieli i położył się do łóżka. Postanowił nie wykonywać skomplikowanej procedury leczenia rany, którą usilnie zalecał mu Osuri. Wczesnym rankiem następnego dnia ugryzienie zamieniło się jednak w ranę martwiczą, wykazującą wyraźne oznaki infekcji.
Niepokojony Iverian aktywował swoje urządzenie Phage-BEAM. BEAM oznaczało “Przyłóżkowy Energetyzowany Środek Przeciwbakteryjny”. Urządzenie miało wielkość i kształt pudełka po butach, ale o bardziej eleganckim i dopracowanym wyglądzie. Nazwa urządzenia i jego producent zostały wypisane kolorowymi literami na boku białego, emaliowanego pudełka. Iverian wyjął wacik ze sterylnego opakowania i delikatnie przejechał nim po całej powierzchni rany, upewniając się, że wysięk z rany dokładnie zwilżył końcówkę wacika. Kiedy wacik zbliżył się do pola “włóż próbkę”, maleńkie drzwiczki otworzyły się jak za dotknięciem czarodziejskiej różdżki, uwalniając wysoki na 10 cali hologram laborantki o imieniu Marcia. Pokazała ona Iverianowi, gdzie należy włożyć próbkę. Marcia została stworzona, aby przeprowadzać klientów przez procedurę testową. “Aby uzyskać najlepsze wyniki, proszę włożyć nowy wkład z bio-inkrustem fagowym, doktorze Iverian” – powiedziała Marcia. Tak jak to było w przypadku drukarek 2D z lat ubiegłych, koszt wkładów z bio-inkrustem był prawie tak wysoki jak koszt samego urządzenia Phage-BEAM. Jak podaje “Business Insider”, bio-ink fagowy był drugim najdroższym płynem na Ziemi, zaraz za Chanel No. 8. Na szczęście, jako jeden z wynalazców, Iverian uzyskał prawo do posiadania zawsze najnowszej wersji tego urządzenia do swojej dyspozycji, w tym ciągłych dostaw odczynników, za darmo.
Iverian doskonale wiedział, jak działa urządzenie, więc nie potrzebował pomocy Marcii. Najpierw z końcówki wacika ekstrahowano DNA i określano metagenom – cały materiał genetyczny obecny w próbce, łącznie z bakteriami zakażającymi. Następnie te dane genetyczne zostały przesłane do zabezpieczonego serwera “Phage XChange”, gdzie złożony algorytm oparty na sztucznej inteligencji przewidział sekwencję genomu faga, który najprawdopodobniej zlizywał bakterie zakażające zidentyfikowane w metagenomie i miał wywołać najsłabszą reakcję immunologiczną u pacjenta. Dane dotyczące genomu faga zostały przesłane do urządzenia Phage-BEAM, które najpierw zsyntetyzowało genom faga, a następnie faga, wykorzystując opatentowany system produkcji fagów bez bakterii.
W ciągu 1 godziny od pobrania próbki urządzenie wyprodukowałoby gotowy do użycia terapeutyczny produkt fagowy. Wyniki procedury krok po kroku byłyby transmitowane na ogromny domowy ekran wideo w salonie Iveriana. Siedząc w swoim fotelu LC2, słuchając Wielkiej Mszy c-moll Mozarta, Iverian z niepokojem oczekiwał na wyniki. Miał co do tego złe przeczucia. Wynik wywołał u Iveriana dreszcze. Zidentyfikowano patogen bakteryjny: Streptococcus pyogenes szczep FE-2033! Osuri natychmiast uruchomił protokół alarmowy, wysyłając wiadomość do Światowego Centrum Kontroli Chorób i podając niepokojące informacje na temat śmiercionośnego, mięsożernego szczepu bakteryjnego, który od 2033 roku był uważany za bezpośrednie zagrożenie dla zdrowia publicznego. Przez chwilę Iverian rozważał wycięcie zainfekowanej rany i kilku otaczających ją zdrowych tkanek nożem kuchennym, ale uspokoił się i postanowił poczekać i zastosować nadchodzący produkt Phage-BEAM. Godzinę później urządzenie Phage-BEAM wytworzyło syntetyczne fagi. Fagi te zostały następnie zmieszane z wyizolowanymi bakteriami, w module walidacyjnym, w celu przetestowania ich skuteczności in vitro. Piętnaście minut później, Iverian otrzymał zielone światło na rozpoczęcie leczenia. Firma Iverian zastosowała fagi w wolno uwalniającym się hydrożelowym opatrunku na ranę, który został uprzednio zmieszany ze stężoną zawiesiną fagów wytworzoną przez urządzenie Phage-BEAM i zawierał również synergistyczne antybiotyki. Hydrożel tymczasowo złagodził ból, co dodatkowo go uspokoiło. Iverian powtarzał aplikację hydrożelu wypełnionego fagami i antybiotykami raz dziennie. Infekcja rany poprawiła się w ciągu 24 godzin, a po 7 dniach rana była prawie całkowicie wyleczona. Potencjalnie zagrażająca życiu infekcja u Iveriana została skutecznie wyleczona, w odpowiednim czasie i bez wychodzenia z domu. Jednak przez wiele poprzednich dekad nie było pewne, czy terapia fagowa przebije się i stanie się szeroko stosowanym i klinicznie użytecznym narzędziem antybakteryjnym. Świat medyczny potrzebował trochę czasu, aby zdać sobie sprawę, że terapia fagowa nie musi być identyczna z antybiotykoterapią, a to głównie z powodu osobliwości czynników aktywnych, fagów.
Niektóre istotne osobliwości fagów
Fagi wykazują szereg właściwości, które różnią się od antybiotyków i utrudniają ich rozwój jako produktów farmaceutycznych i zastosowanie w terapii. Po pierwsze, mają tendencję do bycia specyficznymi w stosunku do bakterii, które infekują. W najlepszym przypadku będą celować w znaczną część jednego gatunku bakterii, ale w najgorszym będą infekować tylko niewielką liczbę szczepów w obrębie jednego gatunku. Fagi terapeutyczne mogą więc być tak dobrane, aby zabijały głównie jeden gatunek bakterii lub jego klinicznie istotną podgrupę, a oszczędzały korzystne dla pacjenta bakterie (np. florę jelitową, skórną lub komensalną w jamie ustnej). Większość rutynowo stosowanych antybiotyków, w przeciwieństwie do nich, ma szerokie spektrum działania, co może powodować “szkody uboczne” w mikrobiomie pacjenta, co z kolei może skutkować działaniami niepożądanymi, takimi jak selekcja gatunków bakterii opornych na antybiotyki (np. Clostridium difficile) lub biegunka związana z antybiotykami (Jernberg et al., 2010). Wadą specyficzności fagów jest to, że przed rozpoczęciem terapii fagowej należy zidentyfikować bakterie zakażające. Z kolei w antybiotykoterapii empirycznej zazwyczaj stosuje się koktajle antybiotyków o szerokim spektrum działania, które działają na wiele bakterii Gram-dodatnich i Gram-ujemnych oraz różnorodne grzyby. Kiedy więcej informacji jest znanych (np. z hodowli bakterii), leczenie może składać się z antybiotyków o wąskim spektrum, które bardziej specyficznie celują w bakterie lub grzyby zidentyfikowane jako powodujące chorobę.
Po drugie, bakterie i fagi są zaangażowane w relacji gospodarz-pasożyt. Ściśle lityczne fagi są wszechobecne w środowisku i wymagają śmierci ich bakteryjnego gospodarza, aby zakończyć ich cykl życiowy. Bez gospodarza fagi nie mogą istnieć. Fagi narzucają selekcję na odpornych gospodarzy, którzy z kolei narzucają selekcję na efektywne fagi. Wynikiem tego jest tak zwana “koewolucja antagonistyczna”, wyścig zbrojeń pomiędzy bakteriami i fagami, charakteryzujący się wzajemną ewolucją oporności bakterii i infekcyjności fagów (Buckling i Rainey, 2002). Podobnie jak w przypadku większości środków przeciwdrobnoustrojowych, bakterie również staną się oporne na fagi (Luria i Delbrück, 1943; Schooley i in., 2017), ale w przeciwieństwie do statycznych antybiotyków, fagi mają zdolność pokonywania oporności bakterii (Buckling i Rainey, 2002). Istnieją jednak przesłanki, że bakterie i fagi nie będą w nieskończoność zwiększać swojej oporności i zakaźności (Fortuna i in., 2019).
Phage Therapy Approaches
W momencie odrodzenia terapii fagowej na początku lat 2000, opracowano dwa odrębne podejścia do terapii fagowej (Pirnay i in., 2011). W podejściu, które można nazwać uniwersalnym, stosowano zdefiniowane koktajle fagowe o szerokim spektrum działania, które miały zwalczać większość bakterii podejrzewanych o wywoływanie określonych chorób zakaźnych. Te predefiniowane koktajle fagowe o szerokim spektrum działania zostały opracowane, wyprodukowane i przetestowane w ramach aktualnych modeli farmakoekonomicznych, które zostały zaprojektowane z myślą o “statycznych” lekach, takich jak antybiotyki. Jednakże, prawdziwie szerokie spektrum działania koktajli fagowych, aktywnych wobec większości bakterii Gram-dodatnich i/lub Gram-ujemnych, powszechnie spotykanych w chorobach zakaźnych, wymagało dużej ilości fagów i okazało się bardzo trudne do opracowania. Możliwe było opracowanie koktajli fagowych o węższym spektrum działania, aktywnych wobec jednego lub kilku gatunków bakterii, które miały być stosowane w określonych wskazaniach i przy założeniu, że zakażające gatunki bakterii były z góry znane. Dla niektórych gatunków bakterii, takich jak Staphylococcus aureus, wyizolowano i scharakteryzowano fagi wykazujące wyjątkowo szeroki zakres gospodarza (Vandersteegen et al., 2011). W PhagoBurn, randomizowanym badaniu kontrolowanym, sukces w osiągnięciu trwałej redukcji obciążenia Pseudomonas aeruginosa w ranach oparzeniowych był związany z początkową wrażliwością na koktajl fagowy (Jault et al., 2019). Wykazano jednak, że jedna trzecia włączonych pacjentów była nosicielami wcześniej istniejących szczepów P. aeruginosa opornych na koktajl fagowy, który składał się z nie mniej niż 12 litycznych fagów P. aeruginosa. Ponadto, koktajle fagowe, które początkowo miały być skuteczne, musiały być regularnie aktualizowane (np. uzupełniane o nowe fagi) w odpowiedzi na pojawienie się oporności fagowej lub zaangażowanie nowo krążących szczepów istotnych klinicznie. Wreszcie, nie było wiadomo, czy konfrontowanie bakterii z wysokimi stężeniami ustalonych koktajli fagowych spowoduje pojawienie się, rozprzestrzenianie i utrzymywanie się oporności bakterii na fagi w szpitalach i w środowisku, podobnie jak to miało miejsce w przypadku masowego stosowania antybiotyków.
W koncepcjach spersonalizowanej terapii fagowej jeden lub więcej fagów wybierano z banku fagów, lub ze środowiska i ewentualnie dostosowywano (selekcja in vitro mutantów fagowych wykazujących zwiększoną infekcyjność) do bardziej efektywnego zakażania bakterii izolowanych z miejsca zakażenia pacjenta (Friman i in., 2016). Niektóre ośrodki terapii fagowej utworzyły i utrzymywały duże banki fagów terapeutycznych, które były regularnie uzupełniane o nowe fagi, poszerzając i dostosowując zakres gospodarza banku do ciągle zmieniających się populacji bakterii. Spersonalizowane metody terapii fagowej były potencjalnie bardziej zrównoważone, ponieważ tylko bakteria infekująca jest celem ataku, co powoduje mniejszą presję selekcyjną w kierunku oporności bakterii na fagi. Jednakże, były one również bardziej skomplikowane i złożone logistycznie niż podejścia uniwersalne, gdzie szczepy bakterii i odpowiadające im fagi były wysyłane na cały świat (rysunek 1). Co więcej, koncepcje medycyny precyzyjnej były, ogólnie rzecz biorąc, niezgodne z większością ścieżek rozwoju i licencjonowania produktów leczniczych (leków w Stanach Zjednoczonych), których ukończenie wymagało kilku lat i milionów euro (dolarów), i to dla każdego faga w banku (Verbeken i in., 2012).
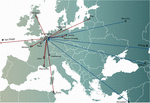
Rysunek 1. Międzynarodowe transfery fagów z (czerwone strzałki) i do (niebieskie strzałki) Szpitala Wojskowego im. Królowej Astrid (QAMH) w Brukseli pod kątem zastosowań klinicznych w latach 2015-2020. Na poziomie krajowym, fagi zostały wysłane z QAMH do pięciu szpitali uniwersyteckich (nie pokazano). Ponadto, wybór pasujących fagów często obejmował transfer izolatów bakteryjnych pacjentów, a pięciu międzynarodowych pacjentów (dwóch z Francji, dwóch z Holandii i jeden z Tunezji) zostało przekazanych do Brukseli w celu terapii fagowej.
Wprowadzenie Biologii Syntetycznej
Z początkiem trzeciego tysiąclecia, podejścia biologii syntetycznej były coraz bardziej rozwijane w celu zmniejszenia specyficzności fagów i pojawienia się oporności bakterii na fagi (np, structure-guided design) (Pires et al., 2016; Dunne et al., 2019). Na przykład, platformy oparte na drożdżach do przełączania białek włókien ogonowych fagów zostały opracowane w celu skonstruowania hybrydowych fagów o bardziej przewidywalnym i rozszerzonym zasięgu gospodarza (Ando i in., 2015; Yosef i in., 2017), a strategie inżynierii genetycznej (np., narzędzia edycji CRISPR-Cas) zostały opracowane w celu rozwiązania innych aspektów, takich jak negatywne interakcje pacjent-fag (np. antyfagowa odpowiedź immunologiczna) (Brown i in., 2017), potencjalne pojawienie się i rozprzestrzenianie mechanizmów oporności fagów bakteryjnych oraz uwalnianie szkodliwych treści bakteryjnych, takich jak endotoksyny (Hwang i in., 2018). Syntetyczne genomy fagowe musiały być restartowane w celu wytworzenia potomstwa fagowego (Barbu i in., 2016; Pires i in., 2016), poprzez transformację form L Escherichia coli lub Listeria monocytogenes (Kilcher i in., 2018) lub przy użyciu bezkomórkowych systemów transkrypcji-translacji (TXTL) (Rustad i in., 2018). Zachodnie ramy regulacyjne stopniowo zaczęły dostosowywać się do precyzyjnych i spersonalizowanych metod terapii fagowej z wykorzystaniem naturalnie występujących fagów (Pirnay i in., 2018), fagów zaprojektowanych (Dedrick i in., 2019) i fagów syntetycznych.
Rozwój urządzeń do produkcji fagów terapeutycznych ad hoc i na miejscu, takich jak Phage-BEAM, nie przebiegał gładko, przypomniał Iverian. Na początek, wymagało to sztucznej inteligencji (AI) opartej na dopasowaniu i projektowaniu fagów in silico. Głębokie uczenie (Martorell-Marugán et al., 2019), podzbiór uczenia maszynowego, zostało wybrane do poszukiwania powiązań między genomami bakterii a genomami infekujących fagów, ponieważ było łatwiejsze do skalowania do większej liczby próbek. Na przykład, metody głębokiego uczenia nie wymagały tzw. ekstrakcji cech, która wymagałaby anotacji na poziomie genów/białek genomów fagów i bakterii oraz ograniczałaby przewidywania do pewnych znanych zależności pomiędzy cechami bakterii i fagów, takich jak struktury włókien ogonowych fagów wiążące się z określonymi receptorami ściany komórkowej bakterii. Wadą tego rozwiązania było to, że musiało być zasilane ciągłym dopływem ogromnej ilości danych, łączących genomy fagów litycznych z genomami bakterii-gospodarzy, i tu właśnie nastąpiło spięcie. Sekwencjonowanie całych genomów powoli przenikało do praktyki mikrobiologii klinicznej (American Academy of Microbiology, 2016), ale instytuty badawcze i firmy farmaceutyczne nie były chętne do przekazywania swoich danych do jednej scentralizowanej bazy danych, nie znaleziono też inwestorów chętnych i zdolnych do pozyskania dostępnych danych i/lub wytworzenia wystarczającej ilości nowych danych. Drugą przeszkodą, którą należało pokonać, była niedostępność szybkiej, niezawodnej i niedrogiej syntezy dużych cząsteczek DNA. Początkowe techniki syntezy DNA opierały się na chemii organicznej i wytwarzały stosunkowo małe cząsteczki DNA. Synteza de novo genomów fagowych wymagała złożenia kilku fragmentów genomu (Barbu i in., 2016; Pires i in., 2016; Lemire i in., 2018) w drożdżach Saccharomyces cerevisiae, przy użyciu sztucznych chromosomów drożdżowych (Ando i in., 2015) lub montażu chemicznego (Gibson i in., 2009). Opracowanie nowej techniki syntezy DNA, opartej na enzymach syntetyzujących DNA, występujących w komórkach układu odpornościowego (Palluk i in., 2018), ułatwiło syntezę genomu fagowego. Wreszcie, trzeba było pokonać pewne przeszkody, aby opracować generyczne systemy bezkomórkowej produkcji fagów zdolne do wytwarzania fagów w wysokich mianach i wykazujących te same poziomy infekcyjności bakterii, co ich naturalne analogi.
Przełom
Głównym problemem było to, że bardzo trudne okazało się zebranie ogromnych ilości powiązanych sekwencji genomów fagowych i bakteryjnych niezbędnych do tego, aby algorytmy głębokiego uczenia AI mogły przewidywać i/lub projektować sekwencje fagów z terapeutycznie akceptowalnym poziomem dokładności. Iverian wspomina, że prawdziwy przełom nastąpił, gdy organizacja non-profit “Phage XChange” uruchomiła swoją globalną platformę zarządzania fagami o tej samej nazwie, aby stworzyć wydajny, standaryzowany, zrównoważony i etyczny łańcuch dostaw fagów (rysunek 2). Phage XChange składa się głównie z modułu AI i księgi rozproszonej (DL) (Thiebes et al., 2020). Moduł AI platformy analizował połączone genomy fagów i bakterii w celu przewidywania i projektowania silnych fagów dla klientów. Przewidywał on również, które patogeny bakteryjne wymagają najpilniejszej uwagi, w oparciu o Internet Rzeczy (IoT) i Big Data oraz informacje dostarczane przez międzynarodowe organizacje zdrowia publicznego, takie jak Światowa Organizacja Zdrowia (WHO) i krajowe Centra Kontroli Chorób. Dane te skierowały system w stronę izolacji i charakterystyki najpilniej potrzebnych fagów.
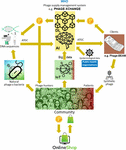
Rysunek 2. Wizja tego, jak łańcuch dostaw fagów może być zorganizowany w 2035 roku. AI, sztuczna inteligencja; ATGC, sekwencja DNA; BEAM, przyłóżkowy energetyzowany antybakteryjny; DL, rozproszona księga rachunkowa; IoT, Internet rzeczy; P, PhageCoin; WHO, Światowa Organizacja Zdrowia.
Moduł DL platformy zapewniał wystarczające, jakościowe i rejestrowane wprowadzanie powiązanych sekwencji genomu fagowego/bakteryjnego do modułu AI i ditto dostarczanie sekwencji fagowych klientom, zgodnie z postanowieniami protokołu z Nagoi (Expert round table on acceptance and re-implementation of bacteriophage therapy et al., 2018). DL w sposób niezmienny rejestrował wszystkich interesariuszy (np. dostawców danych, usług sekwencjonowania i klientów), transakcje i szczegóły umów. Rejestrowała również dokładną jakość, specyfikacje i wagę dostarczonego materiału. Algorytm określał brak redundancji i szacował wagę (np. zjadliwość i zakres gospodarza fagów) oraz przydatność przesłanego materiału. Najbardziej pożądane były oczywiście fagi ukierunkowane na pojawiające się patogeny bakteryjne. Większość kwestii patentowych została pominięta. DL działał jako księga płatności, aby zapewnić, że wszystkie strony zostały zapłacone na czas i sprawiedliwie. Pewna liczba PhageCoins (kryptowaluta platformy) została przypisana dostawcom w zależności od jakości, wagi i atrakcyjności dostarczonego materiału. Klienci wydobywający wyniki predykcji (sekwencje fagów) poprzez DL płacili kwotę PhageCoins, proporcjonalną do szacowanej wartości fagów. Te PhageCoins były używane do utrzymania DL, do zapewnienia wystarczającego i ciągłego dopływu materiału oraz do rozszerzenia danych o zjadliwości fagów i zasięgu gospodarza (dopasowanie fagów do bakterii). Dodatkowy zastrzyk środków i zachętę do dostarczania materiału znaleźliśmy u producentów i dostawców wszelkiego rodzaju dóbr. Za natychmiastowo zarobione PhageCoins dostawcy fagów mogli kupować online wszelkiego rodzaju produkty po bardzo obniżonych cenach, od sprzętu laboratoryjnego i szkolnego po artykuły sportowe. Towary te były dostarczane dzięki sponsorom korporacyjnym. Kilka uznanych firm sponsorowało PhageXchange w zamian za ulgi podatkowe, reklamę i wizerunek marki odpowiedzialnej społecznie. Waga dostarczanego materiału, a więc i jego wartość, były początkowo niedoszacowane, ale były ponownie oceniane w regularnych odstępach czasu (iteracja) i dostawcy otrzymywali więcej PhageCoins, gdy było to uzasadnione. Mimo że platforma była użyteczna od momentu jej wprowadzenia, prawdziwy sukces odniosła dopiero wtedy, gdy została objęta ochroną WHO, analogicznie do światowego systemu identyfikowalności, przejrzystości, czujności i nadzoru nad produktami leczniczymi pochodzenia ludzkiego (Warwick et al., 2013). Formalne porozumienie pomiędzy Phage XChange i WHO zwiększyło międzynarodowe zaufanie do długoterminowej trwałości platformy i ochrony przed nieetycznym wykorzystaniem komercyjnym. Poszukiwanie silnych fagów terapeutycznych szybko stało się wysiłkiem społecznym mającym na celu rozwiązanie kryzysu oporności na antybiotyki, z niezależnymi “łowcami fagów”, szkołami, grupami harcerskimi, wioskami na brzegach Amazonki, itp. izolującymi i przesyłającymi fagi do Phage XChange w zamian za PhageCoins. Na marginesie tego, różne firmy i instytucje opracowały sprzęt i usługi peryferyjne, takie jak zestawy do izolacji fagów oraz platformy do sekwencjonowania i syntezy fagów (np. urządzenie Phage-BEAM). W oczekiwaniu na te urządzenia oferowano rozwiązania pośrednie, w ramach których same fagi były uzyskiwane poprzez DL.
Epilog
Ten pogląd na przyszłość terapii fagowej stanowi optymistyczne zakończenie kryzysu antybiotykooporności. Doraźna i prowadzona na miejscu produkcja syntetycznych fagów, połączona z globalnym, opartym na społeczności, systemem zarządzania fagami, okazała się pożądaną i niedrogą (dla systemów ubezpieczeń społecznych) dodatkową bronią w walce z antybiotykoopornymi infekcjami bakteryjnymi. Nie był to jednak magiczny pocisk, lecz synergiczne uzupełnienie znanych środków przeciwdrobnoustrojowych. Natychmiastowa i bezkomórkowa produkcja syntetycznych fagów, zaprojektowanych lub nie, miała znaczące zalety w porównaniu z klasycznie produkowanymi (w żywicielach bakteryjnych) naturalnymi fagami:
(i) Nie było potrzeby utrzymywania fizycznych banków fagów terapeutycznych i wysyłania izolatów bakteryjnych pacjenta i odpowiadających im fagów terapeutycznych na cały świat.
(ii) Syntetyczne fagi przeciwko bakteriom powodującym poważne zagrożenia dla zdrowia publicznego, takie jak wybuch epidemii E. coli O104:H4 w Niemczech w 2011 roku (Merabishvili i in., 2012), lub bakterie (podejrzane o wykorzystanie) do bioterroryzmu (Jończyk-Matysiak i in., 2014) mogłyby być produkowane na miejscu w odpowiednim czasie.
(iii) Fagi przeciwko bakteriom wywołującym potencjalnie śmiertelne choroby, dla których nie były dostępne szczepy gospodarzy produkcyjnych, które nie są śmiertelne, a których namnażanie wymagało zastosowania środków ostrożności poziomu bezpieczeństwa biologicznego-3 (bsl-3), mogłyby być syntetyzowane w warunkach bsl-1.
(iv) Gdy nie można było wyizolować fagów z miejsc pobierania próbek, na przykład dlatego, że bakteryjne szczepy gospodarza stosowane w technikach izolacji nie były wrażliwe na pożądane fagi, (przewidywane) sekwencje genomowe fagów, wyekstrahowane z danych metagenomicznych (Reyes et al., 2010; Amgarten i in., 2018), mogły być wykorzystane do produkcji syntetycznych fagów.
(v) Preparaty syntetycznych fagów nie zawierały (lub zawierały mniejsze ilości) cząsteczek, które mogłyby mieć negatywny wpływ na pacjentów (np. endotoksyn).
(vi) Urządzenia zostały przystosowane do produkcji syntetycznych fagów podczas przedłużonych podróży kosmicznych i kolonizacji kosmosu (Taylor i Sommer, 2005).
Są małe szanse, że te przewidywania się spełnią. Prawdopodobnie zbyt krótkowzroczne jest myślenie, że wysiłek oparty na społeczności, wspierany przez organizacje zdrowia publicznego i zarządzany przez globalną, zrównoważoną i etyczną platformę, mógłby być sercem rozwiązania obecnego światowego kryzysu oporności na antybiotyki. Niektóre części proponowanego systemu, takie jak bezkomórkowa produkcja syntetycznych fagów za pomocą urządzenia przyłóżkowego, mają rozsądne szanse realizacji, podczas gdy inne elementy, takie jak sponsoring korporacyjny, prawdopodobnie pozostaną ograniczone do sfery fantastyki naukowej. Możesz powiedzieć, że jestem marzycielem, więc nie krępuj się obudzić mnie w 2035 roku, aby skonfrontować mnie z rzeczywistością!
Wkład Autora
J-PP stworzył wizję i sporządził manuskrypt.
Finansowanie
Koszty publikacji zostały pokryte przez “Société Scientifique du Service Médical Militaire – Wetenschappelijke Vereniging van de Militaire Medische Dienst”.
Konflikt interesów
Autor oświadcza, że badania zostały przeprowadzone przy braku jakichkolwiek komercyjnych lub finansowych powiązań, które mogłyby być interpretowane jako potencjalny konflikt interesów.
Redaktor prowadzący zadeklarował współautorstwo z autorem w przeszłości.
Podziękowania
Osobista wizja, czy też marzenie, rozwinięte w tym manuskrypcie powstało w wyniku interakcji z wieloma innymi badaczami w ciągu ostatnich 15 lat. Niemożliwe jest wymienienie ich wszystkich, ale nie byłoby sprawiedliwe, gdybym sam przypisywał sobie wszystkie zasługi. Dlatego zdecydowałem się wymienić tutaj niektórych z nich (w kolejności alfabetycznej), ryzykując – lub lepiej, będąc pewnym – że zapomnę o niektórych ważnych osobach, które wywarły na mnie wpływ: Joana Azeredo, Nata Bakuradze, Bob Blasdel, Dimitri Boeckaerts, Angus Buckling, Yves Briers, Pieter-Jan Ceyssens, Nina Chanishvili, Laurent Debarbieux, Sarah Djebara, Dorien Dams, Daniel De Vos, Quirin Emslander, Alan Fauconnier, Ville Friman, Andrzej Górski, Téa Glonti, Nino Grdzelishvili, Serge Jennes, Elene Kakabadze, Betty Kutter, Rob Lavigne, Cédric Lood, Alice Maestri, Khatuna Makalatia, Maya Merabishvili, Tobi Nagel, Thomas Rose, Patrick Soentjens, Michiel Stock, Rüdiger Trojok, An Van den Bossche, Mario Vaneechoutte, Gilbert Verbeken i Kilian Vogele.
American Academy of Microbiology (2016). Applications of Clinical Microbial Next-Generation Sequencing: Report on an American Academy of Microbiology Colloquium Held in Washington, DC, in April 2015. Washington, DC: American Society for Microbiology.
Google Scholar
Amgarten, D., Braga, L. P. P., da Silva, A. M., and Setubal, J. C. (2018). MARVEL, a tool for prediction of bacteriophage sequences in metagenomic bins. Front. Genet. 9:304. doi: 10.3389/fgene.2018.00304
PubMed Abstract | CrossRef Full Text | Google Scholar
Kilcher, S., Studer, P., Muessner, C., Klumpp, J., and Loessner, M. J. (2018). Cross-genus rebooting of custom-made, synthetic bacteriophage genomes in L-form bacteria. Proc. Natl. Acad. Sci. U.S.A. 115, 567-572. doi: 10.1073/pnas.1714658115
PubMed Abstract | CrossRef Full Text | Google Scholar
Lemire, S., Yehl, K. M., and Lu, T. K. (2018). Aplikacje oparte na fagach w biologii syntetycznej. Annu. Rev. Virol. 5, 453-476. doi: 10.1146/annurev-virology-092917-043544
PubMed Abstract | CrossRef Full Text | Google Scholar
Luria, S. E., and Delbrück, M. (1943). Mutacje bakterii od wrażliwości na wirusy do odporności na wirusy. Genetics 28, 491-511.
Google Scholar
Martorell-Marugán, J., Tabik, S., Benhammou, Y., del Val, C., Zwir, I., Herrera, F., et al. (2019). “Deep learning in omics data analysis and precision medicine,” in Computational Biology, ed. H. Husi (Brisbane: Codon Publications), 37-53. doi: 10.15586/computationalbiology.2019.ch3
PubMed Abstract | CrossRef Full Text | Google Scholar
Merabishvili, M., De Vos, D., Verbeken, G., Kropinski, A. M., Vandenheuvel, D., Lavigne, R., et al. (2012). Selection and characterization of a candidate therapeutic bacteriophage that lyses the Escherichia coli O104:H4 strain from the 2011 outbreak in Germany. PLoS One 7:e52709. doi: 10.1371/journal.pone.0052709
PubMed Abstract | CrossRef Full Text | Google Scholar
Międzybrodzki, R., Borysowski, J., Weber-Da̧browska, B., Fortuna, W., Letkiewicz, S., Szufnarowski, K., et al. (2012). Kliniczne aspekty terapii fagowej. Adv. Virus Res. 83, 73-121. doi: 10.1016/B978-0-12-394438-2.00003-7
PubMed Abstract | CrossRef Full Text | Google Scholar
Palluk, S., Arlow, D. H., de Rond, T., Barthel, S., Kang, J. S., Bector, R., et al. (2018). Synteza DNA de novo z wykorzystaniem koniugatów polimerazy z nukleotydami. Nat. Biotechnol. 36, 645-650. doi: 10.1038/nbt.4173
PubMed Abstract | CrossRef Full Text | Google Scholar
Pires, D. P., Cleto, S., Sillankorva, S., Azeredo, J., and Lu, T. K. (2016). Genetycznie zmodyfikowane fagi: przegląd postępów w ciągu ostatniej dekady. Microbiol. Mol. Biol. Rev. 80, 523-543. doi: 10.1128/MMBR.00069-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., De Vos, D., Verbeken, G., Merabishvili, M., Chanishvili, N., Vaneechoutte, M., et al. (2011). Paradygmat terapii fagowej: prêt-à-porter czy sur-mesure? Pharm. Res. 28, 934-937. doi: 10.1007/s11095-010-0313-5
PubMed Abstract | CrossRef Full Text | Google Scholar
Pirnay, J. P., Verbeken, G., Ceyssens, P. J., Huys, I., De Vos, D., Ameloot, C., et al. (2018). The magistral phage. Viruses 10:E64. doi: 10.3390/v10020064
PubMed Abstract | CrossRef Full Text | Google Scholar
Reyes, A., Haynes, M., Hanson, N., Angly, F. E., Heath, A. C., Rohwer, F., et al. (2010). Viruses in the fecal microbiota of monozygotic twins and their mothers. Nature 466, 334-338. doi: 10.1038/nature09199
PubMed Abstract | CrossRef Full Text | Google Scholar
Rustad, M., Eastlund, A., Jardine, P., and Noireaux, V. (2018). Bezkomórkowa synteza TXTL zakaźnego bakteriofaga T4 w pojedynczej reakcji w probówce. Synth. Biol. 3:ysy002.
Google Scholar
Schooley, R. T., Biswas, B., Gill, J. J., Hernandez-Morales, A., Lancaster, J., Lessor, L., et al. (2017). Opracowanie i zastosowanie spersonalizowanych koktajli terapeutycznych opartych na bakteriofagach w leczeniu pacjenta z rozsianą oporną infekcją Acinetobacter baumannii. Antimicrob. Agents Chemother. 61:e0954-17. doi: 10.1128/AAC.02221-18
PubMed Abstract | CrossRef Full Text | Google Scholar
Sulakvelidze, A., Alavidze, Z., and Morris, J. G. Jr. (2001). Bacteriophage therapy. Antimicrob. Agents Chemother. 45, 649-659.
Google Scholar
Suttle, C. A. (2013). Wirusy: odblokowywanie największej bioróżnorodności na Ziemi. Genome 56, 542-544. doi: 10.1139/gen-2013-0152
PubMed Abstract | CrossRef Full Text | Google Scholar
Taylor, P. W., and Sommer, A. P. (2005). Towards rational treatment of bacterial infections during extended space travel. Int. J. Antimicrob. Agents 26, 183-187. doi: 10.1016/j.ijantimicag.2005.06.002
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiebes, S., Schlesner, M., Brors, B., and Sunyaev, A. (2020). Distributed ledger technology in genomics: a call for Europe. Eur. J. Hum. Genet. 28, 139-140. doi: 10.1038/s41431-019-0512-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Thiel, K. (2004). Stary dogmat, nowe sztuczki – terapia fagowa XXI wieku. Nat. Biotechnol. 2, 31-36. doi: 10.1038/nbt0104-31
PubMed Abstract | CrossRef Full Text | Google Scholar
Vandersteegen, K., Mattheus, W., Ceyssens, P. J., Bilocq, F., De Vos, D., Pirnay, J.-P., et al. (2011). Microbiological and molecular assessment of bacteriophage ISP for the control of Staphylococcus aureus. PLoS One 6:e24418. doi: 10.1371/journal.pone.0024418
PubMed Abstract | CrossRef Full Text | Google Scholar
Verbeken, G., Pirnay, J. P., De Vos, D., Jennes, S., Zizi, M., Lavigne, R., et al. (2012). Optimizing the European regulatory framework for sustainable bacteriophage therapy in human medicine. Arch. Immunol. Ther. Exp. 60, 161-172. doi: 10.1007/s00005-012-0175-0
PubMed Abstract | CrossRef Full Text | Google Scholar
Warwick, R. M., Chapman, J., Pruett, T. L., and Wang, H. (2013). Globally consistent coding systems for medical products of human origin. Bull. World Health Organ. 91, 314-314A. doi: 10.2471/BLT.12.116988
PubMed Abstract | CrossRef Full Text | Google Scholar
Yosef, I., Goren, M. G., Globus, R., Molshanski-Mor, S., and Qimron, U. (2017). Extending the host range of bacteriophage particles for DNA transduction. Mol. Cell. 66, 721.e3-728.e3. doi: 10.1016/j.molcel.2017.04.025
PubMed Abstract | CrossRef Full Text | Google Scholar
.